for (pkg in c("tidyverse")) {
if (!require(pkg, character.only = TRUE)) install.packages(pkg)
}
library(tidyverse)Um alle in diesem Kapitel verwendeten Pakete zu installieren und zu laden, führt man folgenden Code aus:
Einleitung
In der Praxis liegen Daten selten in einer einzigen, perfekt aufbereiteten Tabelle vor. Stattdessen hat man oft mehrere Datenquellen, die man zusammenführen muss: Messwerte aus verschiedenen Laboren, Stammdaten und Transaktionsdaten, oder einfach Daten, die über mehrere Excel-Sheets verteilt sind. Dieses Kapitel zeigt, wie man in R Tabellen auf verschiedene Arten kombinieren kann.
Wir unterscheiden dabei drei grundlegende Ansätze:
-
Stapeln: Tabellen einfach untereinander (
bind_rows()) oder nebeneinander (bind_cols()) zusammenfügen - Joinen: Tabellen anhand gemeinsamer Schlüsselspalten intelligent verknüpfen
- Umstrukturieren: Daten zwischen “breitem” und “langem” Format transformieren
Tabellen stapeln
Die einfachste Art, Tabellen zu kombinieren, ist das “Stapeln” - also Tabellen entweder untereinander oder nebeneinander zusammenzufügen. Hierfür gibt es bind_rows() und bind_cols().
Beispieldaten
Für diesen Abschnitt erstellen wir drei kleine Tibbles mit Obstdaten:
obst_1# A tibble: 2 × 2
sorte preis
<chr> <dbl>
1 Apfel 1.2
2 Birne 1.5obst_2# A tibble: 2 × 2
sorte preis
<chr> <dbl>
1 Orange 0.8
2 Banane 1.1obst_3# A tibble: 2 × 3
sorte preis herkunft
<chr> <dbl> <chr>
1 Kirsche 3.5 Deutschland
2 Pflaume 2.2 Spanien Man beachte: obst_1 und obst_2 haben dieselben Spalten (sorte und preis), während obst_3 eine zusätzliche Spalte herkunft hat.
bind_rows()
Die Funktion bind_rows() stapelt Tabellen untereinander - sie fügt also Zeilen hinzu. Das ist nützlich, wenn man z.B. Daten aus verschiedenen Zeiträumen oder verschiedenen Quellen hat, die dieselbe Struktur haben.
bind_rows(obst_1, obst_2)# A tibble: 4 × 2
sorte preis
<chr> <dbl>
1 Apfel 1.2
2 Birne 1.5
3 Orange 0.8
4 Banane 1.1Das funktioniert wie erwartet: Die Zeilen werden einfach untereinander gehängt.
Unterschiedliche Spalten
Der große Vorteil von bind_rows() gegenüber der base-R-Funktion rbind() zeigt sich, wenn die Tabellen unterschiedliche Spalten haben. Während rbind() in diesem Fall einen Fehler wirft, fügt bind_rows() die Tabellen trotzdem zusammen und füllt fehlende Werte mit NA:
bind_rows(obst_1, obst_3)# A tibble: 4 × 3
sorte preis herkunft
<chr> <dbl> <chr>
1 Apfel 1.2 <NA>
2 Birne 1.5 <NA>
3 Kirsche 3.5 Deutschland
4 Pflaume 2.2 Spanien Man sieht: obst_1 hatte keine herkunft-Spalte, also werden diese Werte mit NA aufgefüllt. Das ist sehr praktisch, wenn man Daten aus verschiedenen Quellen kombiniert, die nicht exakt dieselben Spalten haben.
Herkunft markieren mit .id
Wenn man mehrere Tabellen zusammenfügt, möchte man oft wissen, aus welcher Ursprungstabelle jede Zeile stammt. Dafür gibt es das .id-Argument:
bind_rows(
"Laden_A" = obst_1,
"Laden_B" = obst_2,
.id = "quelle"
)# A tibble: 4 × 3
quelle sorte preis
<chr> <chr> <dbl>
1 Laden_A Apfel 1.2
2 Laden_A Birne 1.5
3 Laden_B Orange 0.8
4 Laden_B Banane 1.1Hier haben wir den Tabellen Namen gegeben (“Laden_A”, “Laden_B”) und mit .id = "quelle" eine neue Spalte erstellt, die diese Namen enthält.
Alle drei Tabellen kombinieren
Man kann auch mehr als zwei Tabellen auf einmal stapeln:
bind_rows(obst_1, obst_2, obst_3)# A tibble: 6 × 3
sorte preis herkunft
<chr> <dbl> <chr>
1 Apfel 1.2 <NA>
2 Birne 1.5 <NA>
3 Orange 0.8 <NA>
4 Banane 1.1 <NA>
5 Kirsche 3.5 Deutschland
6 Pflaume 2.2 Spanien Die Spalte herkunft existiert nur für die letzten zwei Zeilen (aus obst_3), alle anderen bekommen NA.
bind_cols()
Die Funktion bind_cols() fügt Tabellen nebeneinander zusammen - sie klebt also Spalten aneinander.
Bei bind_cols() gibt es keine intelligente Verknüpfung über Schlüsselspalten! Die Tabellen werden einfach “blind” nebeneinander geklebt. Das bedeutet: Die Zeilen müssen in exakt derselben Reihenfolge stehen, und die Tabellen müssen gleich viele Zeilen haben.
Ein Beispiel:
# A tibble: 3 × 4
vorname nachname alter beruf
<chr> <chr> <dbl> <chr>
1 Anna Mueller 28 Aerztin
2 Ben Schmidt 34 Ingenieur
3 Clara Weber 22 StudentinDas funktioniert, weil beide Tibbles drei Zeilen haben und wir wissen, dass Zeile 1 in beiden Tibbles zur selben Person gehört.
Wann ist bind_cols() gefährlich?
bind_cols() kann zu falschen Ergebnissen führen, wenn die Reihenfolge der Zeilen nicht übereinstimmt:
# A tibble: 3 × 4
vorname nachname alter beruf
<chr> <chr> <dbl> <chr>
1 Anna Mueller 28 Aerztin
2 Ben Schmidt 34 Ingenieur
3 Clara Weber 22 StudentinHier wurden die Namen alphabetisch sortiert, aber die Alter-Daten nicht - Anna bekommt jetzt das Alter 28 zugewiesen, obwohl das eigentlich zu “Anna Mueller” vor der Sortierung gehörte (und jetzt zufällig stimmt, aber Ben und Clara sind vertauscht!). Das ist ein häufiger Fehler!
Wann sollte man bind_cols() verwenden?
bind_cols() ist sicher, wenn:
- Die Daten aus derselben Quelle stammen und garantiert dieselbe Reihenfolge haben
- Man gerade selbst mehrere Berechnungen auf denselben Daten durchgeführt hat
- Man nach dem Zusammenfügen die Korrektheit überprüft
In den meisten anderen Fällen ist ein Join die bessere Wahl, weil dort über eine Schlüsselspalte verknüpft wird.
Tabellen joinen
Joins sind die mächtigste Methode, um Tabellen zu kombinieren. Sie verknüpfen Tabellen intelligent über eine oder mehrere gemeinsame Spalten (die “Schlüssel” oder “Keys”). Dadurch ist es egal, in welcher Reihenfolge die Zeilen stehen - R findet die zusammengehörigen Zeilen automatisch.
Beispieldaten
Für die Joins verwenden wir einen anderen Datensatz: Städtedaten. Wir erstellen drei Tibbles mit unterschiedlichen Informationen über Städte:
# Tibble 1: Sechs Grossstaedte in Zentraleuropa mit Einwohnerzahlen
staedte_europa <- tibble(
stadt = c("Berlin", "Hamburg", "Muenchen", "Kopenhagen", "Amsterdam", "London"),
einwohner_mio = c(3.9, 1.9, 1.5, 0.7, 0.9, 9.0)
)
# Tibble 2: Zehn deutsche Staedte mit Mietpreisen (Euro pro Quadratmeter)
staedte_miete <- tibble(
stadt = c("Berlin", "Hamburg", "Muenchen", "Frankfurt", "Koeln",
"Duesseldorf", "Stuttgart", "Leipzig", "Dresden", "Nuernberg"),
miete_qm = c(18.29, 17.18, 22.64, 19.62, 15.21,
16.04, 17.26, 11.38, 7.33, 9.65)
)
# Tibble 3: Dieselben zehn deutschen Staedte mit weiteren Statistiken
staedte_stats <- tibble(
stadt = c("Berlin", "Hamburg", "Muenchen", "Frankfurt", "Koeln",
"Duesseldorf", "Stuttgart", "Leipzig", "Dresden", "Nuernberg"),
flaeche_km2 = c(892, 755, 310, 248, 405, 217, 207, 297, 328, 186),
gruenflaeche_pct = c(14.4, 16.8, 11.9, 21.5, 17.2, 18.9, 24.0, 14.8, 12.3, 19.1)
)staedte_europa# A tibble: 6 × 2
stadt einwohner_mio
<chr> <dbl>
1 Berlin 3.9
2 Hamburg 1.9
3 Muenchen 1.5
4 Kopenhagen 0.7
5 Amsterdam 0.9
6 London 9 staedte_miete# A tibble: 10 × 2
stadt miete_qm
<chr> <dbl>
1 Berlin 18.3
2 Hamburg 17.2
3 Muenchen 22.6
4 Frankfurt 19.6
5 Koeln 15.2
6 Duesseldorf 16.0
7 Stuttgart 17.3
8 Leipzig 11.4
9 Dresden 7.33
10 Nuernberg 9.65staedte_stats# A tibble: 10 × 3
stadt flaeche_km2 gruenflaeche_pct
<chr> <dbl> <dbl>
1 Berlin 892 14.4
2 Hamburg 755 16.8
3 Muenchen 310 11.9
4 Frankfurt 248 21.5
5 Koeln 405 17.2
6 Duesseldorf 217 18.9
7 Stuttgart 207 24
8 Leipzig 297 14.8
9 Dresden 328 12.3
10 Nuernberg 186 19.1Man beachte: staedte_europa enthält drei deutsche Städte (Berlin, Hamburg, Muenchen), die auch in den anderen beiden Tibbles vorkommen, plus drei nicht-deutsche Städte. Die Tibbles staedte_miete und staedte_stats haben exakt dieselben zehn deutschen Städte, aber unterschiedliche Spalten.
Das Konzept: Schlüsselspalten
Bei einem Join gibt man an, welche Spalte(n) als “Schlüssel” verwendet werden sollen. R sucht dann nach übereinstimmenden Werten in dieser Spalte und fügt die entsprechenden Zeilen zusammen.
In unseren Beispieldaten ist stadt die offensichtliche Schlüsselspalte - sie kommt in allen drei Tibbles vor und identifiziert eindeutig jede Zeile.
Mutating Joins
“Mutating Joins” fügen Spalten aus einer Tabelle zu einer anderen hinzu - sie “mutieren” also die Ausgangstabelle, indem sie sie um neue Spalten erweitern. Es gibt vier Varianten, die sich darin unterscheiden, welche Zeilen im Ergebnis enthalten sind.
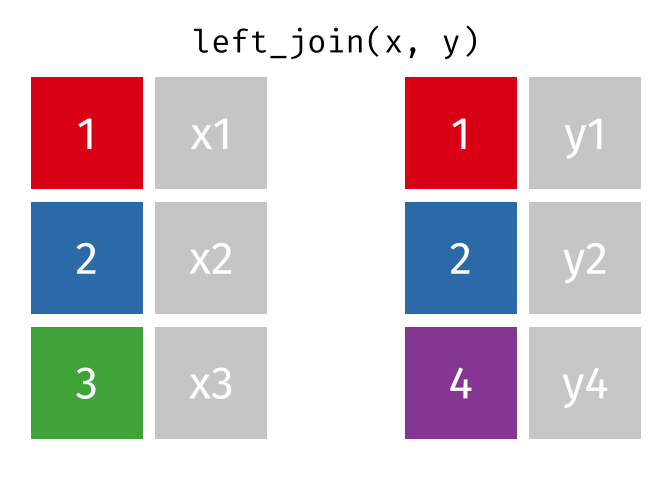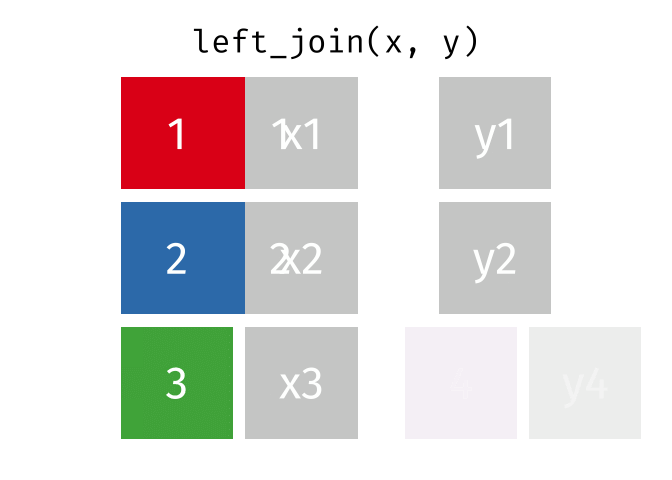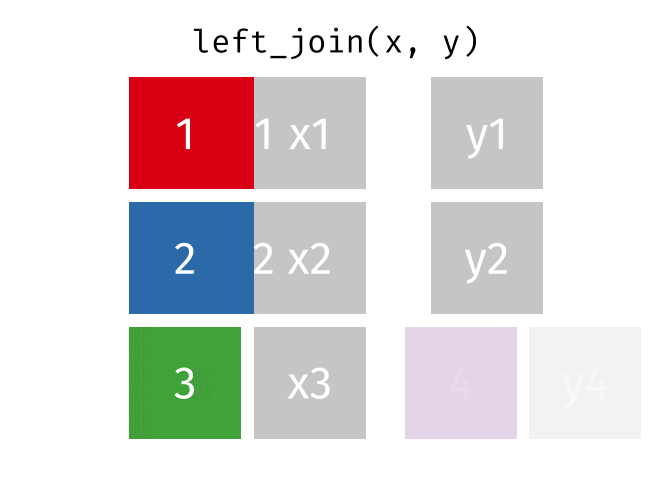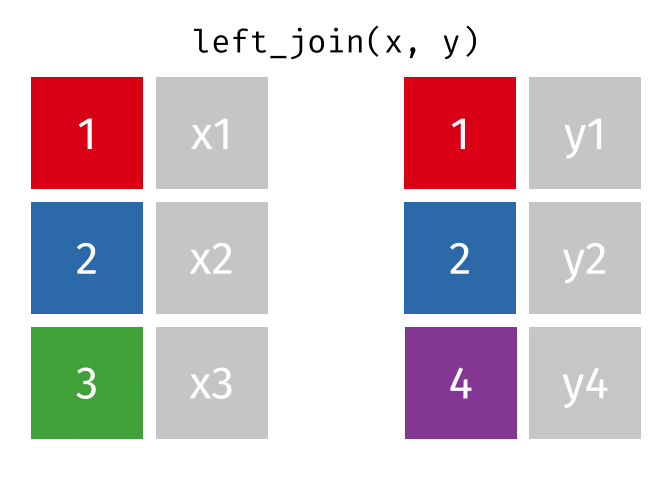
left_join()
Der left_join() behält alle Zeilen aus der linken Tabelle und fügt passende Spalten aus der rechten Tabelle hinzu. Wenn es keinen passenden Partner in der rechten Tabelle gibt, werden die neuen Spalten mit NA gefüllt.
Die animierten Grafiken in diesem Kapitel stammen von Garrick Aden-Buie. Er hat dort eine fantastische Sammlung von Visualisierungen erstellt, die die verschiedenen Join-Typen und andere tidyverse-Operationen veranschaulichen. Ein Besuch lohnt sich!
# A tibble: 6 × 3
stadt einwohner_mio miete_qm
<chr> <dbl> <dbl>
1 Berlin 3.9 18.3
2 Hamburg 1.9 17.2
3 Muenchen 1.5 22.6
4 Kopenhagen 0.7 NA
5 Amsterdam 0.9 NA
6 London 9 NA Man sieht:
- Alle 6 Städte aus
staedte_europasind im Ergebnis - Berlin, Hamburg und Muenchen haben Mietpreise bekommen
- Kopenhagen, Amsterdam und London haben
NAbeimiete_qm, weil sie nicht instaedte_mietevorkommen
Der left_join() ist der am häufigsten verwendete Join, weil man oft eine “Haupttabelle” hat, die man um zusätzliche Informationen erweitern möchte, ohne Zeilen zu verlieren.
right_join()
Der right_join() ist das Spiegelbild des left_join(): Er behält alle Zeilen aus der rechten Tabelle.
staedte_europa %>%
right_join(staedte_miete, by = "stadt")# A tibble: 10 × 3
stadt einwohner_mio miete_qm
<chr> <dbl> <dbl>
1 Berlin 3.9 18.3
2 Hamburg 1.9 17.2
3 Muenchen 1.5 22.6
4 Frankfurt NA 19.6
5 Koeln NA 15.2
6 Duesseldorf NA 16.0
7 Stuttgart NA 17.3
8 Leipzig NA 11.4
9 Dresden NA 7.33
10 Nuernberg NA 9.65Jetzt haben wir:
- Alle 10 deutschen Städte aus
staedte_miete - Berlin, Hamburg und Muenchen haben Einwohnerzahlen
- Die 7 anderen deutschen Städte haben
NAbeieinwohner_mio
In der Praxis kann man statt right_join(a, b) auch einfach left_join(b, a) schreiben - das Ergebnis ist dasselbe (nur die Spaltenreihenfolge unterscheidet sich). Viele R-Nutzer verwenden daher fast ausschließlich left_join().
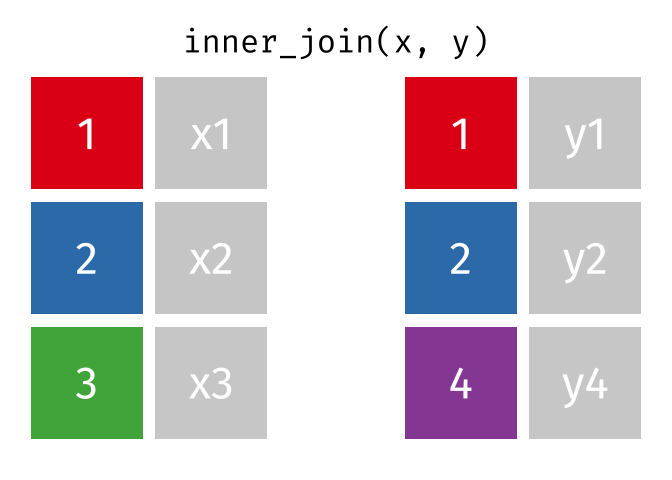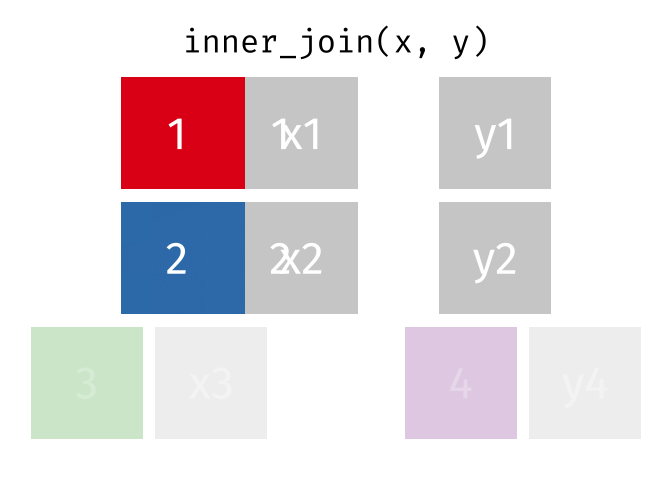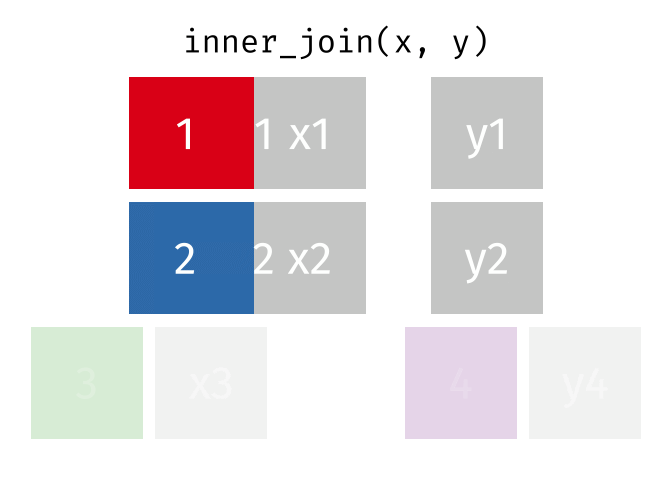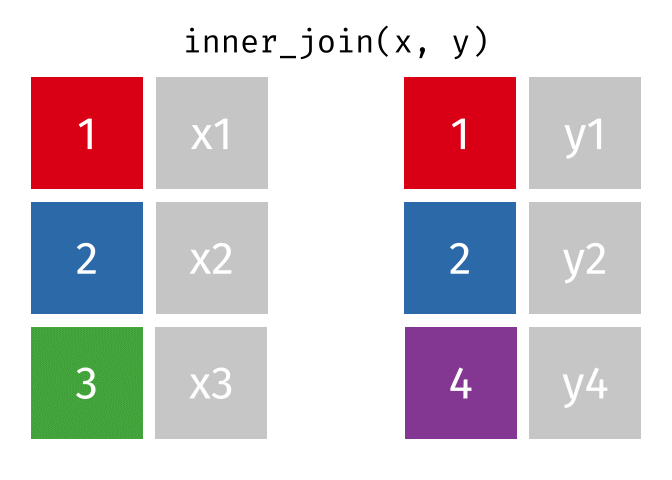
inner_join()
Der inner_join() behält nur Zeilen, die in beiden Tabellen vorkommen. Zeilen ohne Partner werden komplett ausgeschlossen.
staedte_europa %>%
inner_join(staedte_miete, by = "stadt")# A tibble: 3 × 3
stadt einwohner_mio miete_qm
<chr> <dbl> <dbl>
1 Berlin 3.9 18.3
2 Hamburg 1.9 17.2
3 Muenchen 1.5 22.6Nur Berlin, Hamburg und Muenchen sind übrig - die einzigen Städte, die in beiden Tabellen vorkommen. Es gibt keine NA-Werte im Ergebnis.
full_join()
Der full_join() behält alle Zeilen aus beiden Tabellen. Das ist die “großzügigste” Variante.
# A tibble: 13 × 3
stadt einwohner_mio miete_qm
<chr> <dbl> <dbl>
1 Berlin 3.9 18.3
2 Hamburg 1.9 17.2
3 Muenchen 1.5 22.6
4 Kopenhagen 0.7 NA
5 Amsterdam 0.9 NA
6 London 9 NA
7 Frankfurt NA 19.6
8 Koeln NA 15.2
9 Duesseldorf NA 16.0
10 Stuttgart NA 17.3
11 Leipzig NA 11.4
12 Dresden NA 7.33
13 Nuernberg NA 9.65Das Ergebnis hat 13 Zeilen: 3 deutsche Städte mit vollständigen Daten, 3 nicht-deutsche Städte (nur Einwohner), und 7 weitere deutsche Städte (nur Miete).
Übung: Joins mit Pflanzendaten
Bereite zunächst die Daten vor:
# PlantGrowth-Datensatz laden und erweitern
data(PlantGrowth)
# Datensatz 1: Gewichtsmessungen mit eindeutiger ID
pflanzen_gewicht <- PlantGrowth %>%
mutate(plant_id = 1:n()) %>%
select(plant_id, group, weight)
# Datensatz 2: Höhenmessungen (nur für einen Teil der Pflanzen verfügbar!)
set.seed(123)
pflanzen_hoehe <- tibble(
plant_id = c(1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29),
hoehe_cm = round(rnorm(15, mean = 26, sd = 3), 1)
)
# Die Datensätze anschauen
pflanzen_gewicht plant_id group weight
1 1 ctrl 4.17
2 2 ctrl 5.58
3 3 ctrl 5.18
4 4 ctrl 6.11
5 5 ctrl 4.50
6 6 ctrl 4.61
7 7 ctrl 5.17
8 8 ctrl 4.53
9 9 ctrl 5.33
10 10 ctrl 5.14
11 11 trt1 4.81
12 12 trt1 4.17
13 13 trt1 4.41
14 14 trt1 3.59
15 15 trt1 5.87
16 16 trt1 3.83
17 17 trt1 6.03
18 18 trt1 4.89
19 19 trt1 4.32
20 20 trt1 4.69
21 21 trt2 6.31
22 22 trt2 5.12
23 23 trt2 5.54
24 24 trt2 5.50
25 25 trt2 5.37
26 26 trt2 5.29
27 27 trt2 4.92
28 28 trt2 6.15
29 29 trt2 5.80
30 30 trt2 5.26pflanzen_hoehe# A tibble: 15 × 2
plant_id hoehe_cm
<dbl> <dbl>
1 1 24.3
2 3 25.3
3 5 30.7
4 7 26.2
5 9 26.4
6 11 31.1
7 13 27.4
8 15 22.2
9 17 23.9
10 19 24.7
11 21 29.7
12 23 27.1
13 25 27.2
14 27 26.3
15 29 24.3Beantworte die folgenden Fragen mithilfe der passenden Join-Funktionen:
Füge die Höhenmessungen zu allen Pflanzen hinzu. Pflanzen ohne Höhenmessung sollen
NAbekommen. Wie viele Pflanzen haben eine Höhenmessung?Erstelle einen Datensatz mit nur den Pflanzen, für die sowohl Gewicht als auch Höhe gemessen wurden.
Welche Pflanzen (plant_id) haben keine Höhenmessung? Nutze einen Filtering Join.
Berechne für die Pflanzen mit beiden Messungen das Verhältnis
weight / hoehe_cmund speichere es in einer neuen Spalteratio.
plant_id group weight hoehe_cm
1 1 ctrl 4.17 24.3
2 2 ctrl 5.58 NA
3 3 ctrl 5.18 25.3
4 4 ctrl 6.11 NA
5 5 ctrl 4.50 30.7
6 6 ctrl 4.61 NA
7 7 ctrl 5.17 26.2
8 8 ctrl 4.53 NA
9 9 ctrl 5.33 26.4
10 10 ctrl 5.14 NA
11 11 trt1 4.81 31.1
12 12 trt1 4.17 NA
13 13 trt1 4.41 27.4
14 14 trt1 3.59 NA
15 15 trt1 5.87 22.2
16 16 trt1 3.83 NA
17 17 trt1 6.03 23.9
18 18 trt1 4.89 NA
19 19 trt1 4.32 24.7
20 20 trt1 4.69 NA
21 21 trt2 6.31 29.7
22 22 trt2 5.12 NA
23 23 trt2 5.54 27.1
24 24 trt2 5.50 NA
25 25 trt2 5.37 27.2
26 26 trt2 5.29 NA
27 27 trt2 4.92 26.3
28 28 trt2 6.15 NA
29 29 trt2 5.80 24.3
30 30 trt2 5.26 NA[1] 15# b) inner_join: Nur Pflanzen mit beiden Messungen
pflanzen_beide <- pflanzen_gewicht %>%
inner_join(pflanzen_hoehe, by = "plant_id")
pflanzen_beide plant_id group weight hoehe_cm
1 1 ctrl 4.17 24.3
2 3 ctrl 5.18 25.3
3 5 ctrl 4.50 30.7
4 7 ctrl 5.17 26.2
5 9 ctrl 5.33 26.4
6 11 trt1 4.81 31.1
7 13 trt1 4.41 27.4
8 15 trt1 5.87 22.2
9 17 trt1 6.03 23.9
10 19 trt1 4.32 24.7
11 21 trt2 6.31 29.7
12 23 trt2 5.54 27.1
13 25 trt2 5.37 27.2
14 27 trt2 4.92 26.3
15 29 trt2 5.80 24.3 plant_id group weight
1 2 ctrl 5.58
2 4 ctrl 6.11
3 6 ctrl 4.61
4 8 ctrl 4.53
5 10 ctrl 5.14
6 12 trt1 4.17
7 14 trt1 3.59
8 16 trt1 3.83
9 18 trt1 4.89
10 20 trt1 4.69
11 22 trt2 5.12
12 24 trt2 5.50
13 26 trt2 5.29
14 28 trt2 6.15
15 30 trt2 5.26 plant_id group weight hoehe_cm ratio
1 1 ctrl 4.17 24.3 0.1716049
2 3 ctrl 5.18 25.3 0.2047431
3 5 ctrl 4.50 30.7 0.1465798
4 7 ctrl 5.17 26.2 0.1973282
5 9 ctrl 5.33 26.4 0.2018939
6 11 trt1 4.81 31.1 0.1546624
7 13 trt1 4.41 27.4 0.1609489
8 15 trt1 5.87 22.2 0.2644144
9 17 trt1 6.03 23.9 0.2523013
10 19 trt1 4.32 24.7 0.1748988
11 21 trt2 6.31 29.7 0.2124579
12 23 trt2 5.54 27.1 0.2044280
13 25 trt2 5.37 27.2 0.1974265
14 27 trt2 4.92 26.3 0.1870722
15 29 trt2 5.80 24.3 0.2386831Unterschiedliche Spaltennamen
Manchmal heißt die Schlüsselspalte in den beiden Tabellen unterschiedlich. Dann kann man das im by-Argument angeben:
# A tibble: 10 × 3
stadt miete_qm population
<chr> <dbl> <dbl>
1 Berlin 18.3 3.8
2 Hamburg 17.2 1.9
3 Muenchen 22.6 1.5
4 Frankfurt 19.6 NA
5 Koeln 15.2 NA
6 Duesseldorf 16.0 NA
7 Stuttgart 17.3 NA
8 Leipzig 11.4 NA
9 Dresden 7.33 NA
10 Nuernberg 9.65 NA Die Syntax by = c("stadt" = "city") bedeutet: “Verknüpfe die Spalte stadt aus der linken Tabelle mit der Spalte city aus der rechten Tabelle.”
Filtering Joins
Im Gegensatz zu den Mutating Joins fügen Filtering Joins keine neuen Spalten hinzu. Sie filtern nur die Zeilen der linken Tabelle basierend darauf, ob es einen Partner in der rechten Tabelle gibt.
semi_join()
Der semi_join() behält alle Zeilen aus der linken Tabelle, die einen Partner in der rechten Tabelle haben.
# A tibble: 3 × 2
stadt einwohner_mio
<chr> <dbl>
1 Berlin 3.9
2 Hamburg 1.9
3 Muenchen 1.5Das Ergebnis enthält nur Berlin, Hamburg und Muenchen - die europäischen Städte, für die wir Mietdaten haben. Aber: Es wurden keine Spalten aus staedte_miete hinzugefügt! Das Ergebnis hat nur die Spalten von staedte_europa.
Der semi_join() beantwortet die Frage: “Welche Zeilen aus Tabelle A haben einen Partner in Tabelle B?”
anti_join()
Der anti_join() ist das Gegenteil: Er behält alle Zeilen aus der linken Tabelle, die keinen Partner in der rechten Tabelle haben.
# A tibble: 3 × 2
stadt einwohner_mio
<chr> <dbl>
1 Kopenhagen 0.7
2 Amsterdam 0.9
3 London 9 Kopenhagen, Amsterdam und London - die europäischen Städte, für die wir keine Mietdaten haben.
Der anti_join() ist sehr nützlich zur Datenqualitätsprüfung: “Welche Datensätze fehlen?” oder “Welche IDs aus System A gibt es nicht in System B?”
Set Operations
Set Operations behandeln Tabellen wie mathematische Mengen. Sie funktionieren nur, wenn beide Tabellen exakt dieselben Spalten haben. Sie vergleichen dann ganze Zeilen (nicht einzelne Schlüsselspalten).
Für die Beispiele erstellen wir zwei kleine Tabellen mit identischen Spalten:
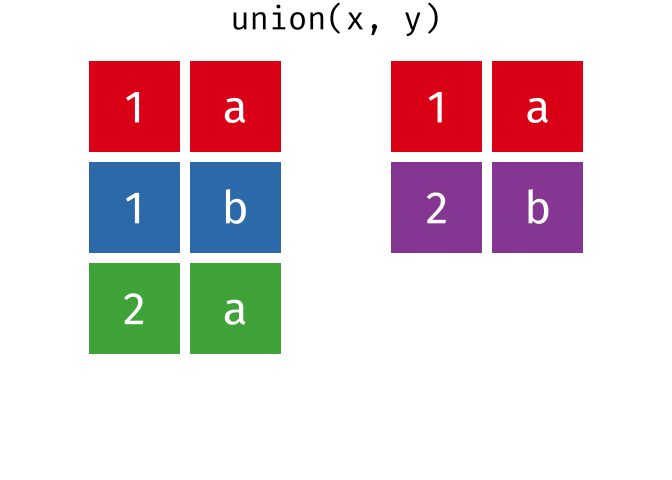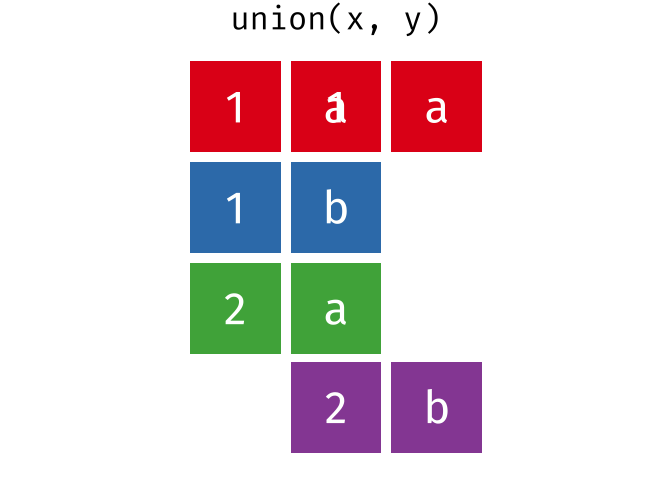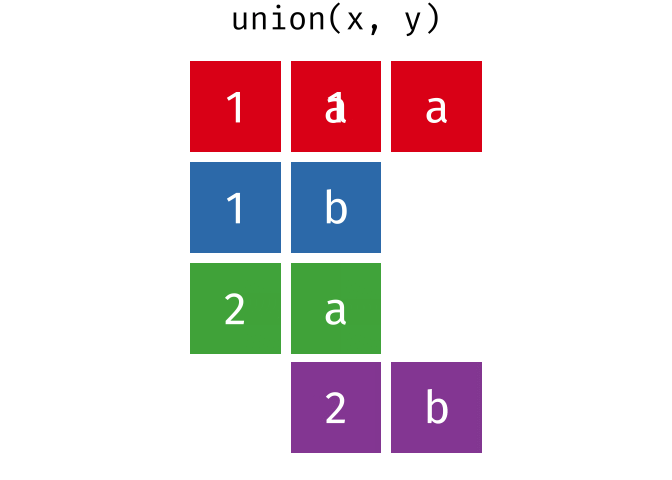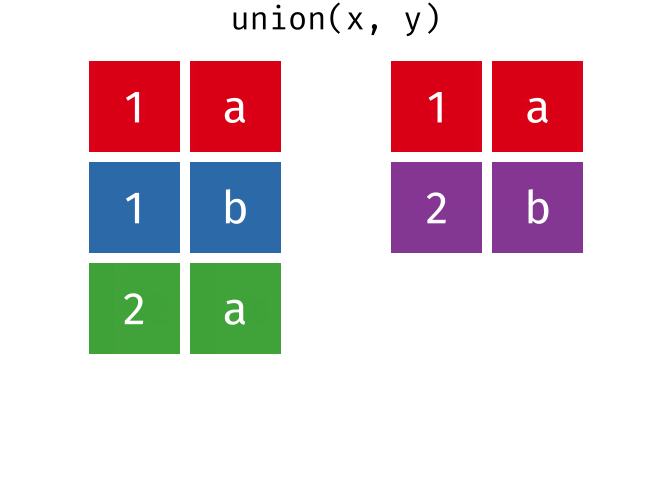
union()
union() gibt alle einzigartigen Zeilen aus beiden Tabellen zurück - also die Vereinigungsmenge.
union(menge_a, menge_b)# A tibble: 4 × 2
stadt land
<chr> <chr>
1 Berlin Deutschland
2 Hamburg Deutschland
3 Muenchen Deutschland
4 Frankfurt DeutschlandHamburg und Muenchen kommen in beiden Tabellen vor, erscheinen im Ergebnis aber nur einmal.
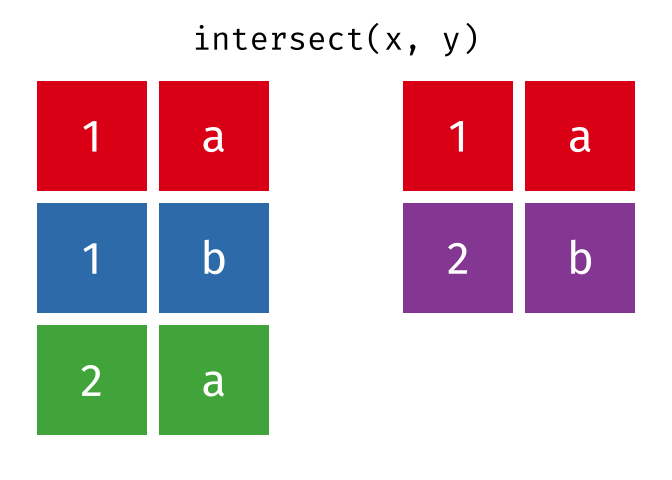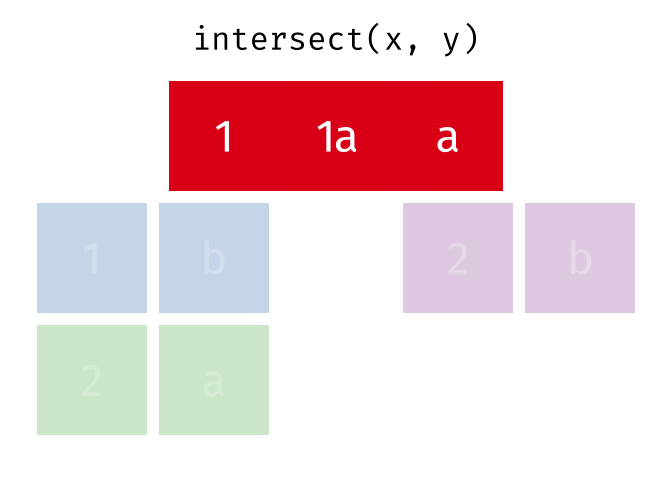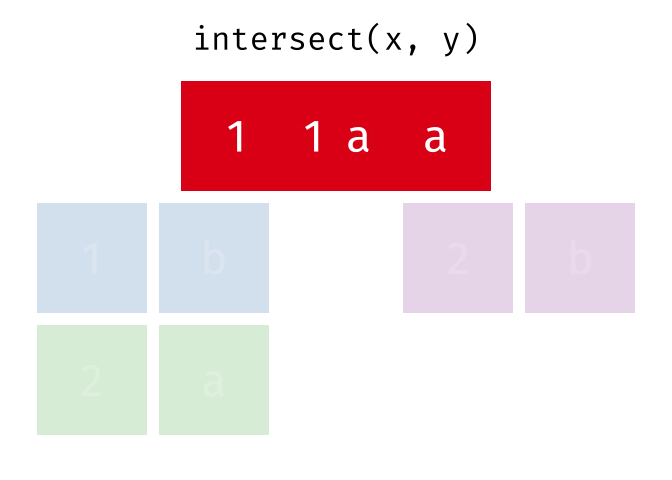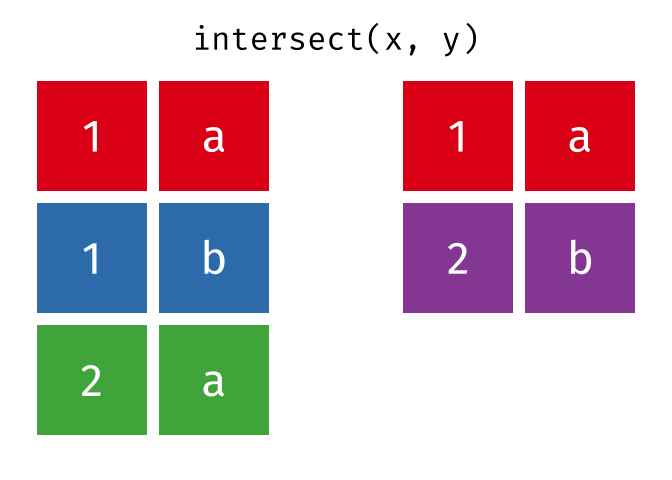
intersect()
intersect() gibt nur die Zeilen zurück, die in beiden Tabellen vorkommen - also die Schnittmenge.
intersect(menge_a, menge_b)# A tibble: 2 × 2
stadt land
<chr> <chr>
1 Hamburg Deutschland
2 Muenchen DeutschlandNur Hamburg und Muenchen sind in beiden Tabellen.
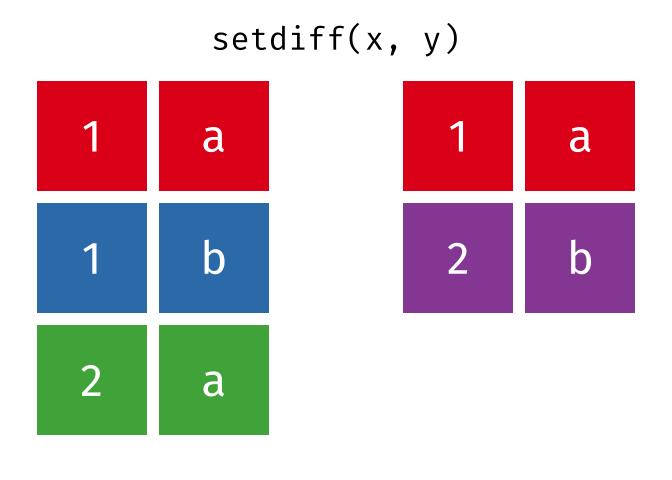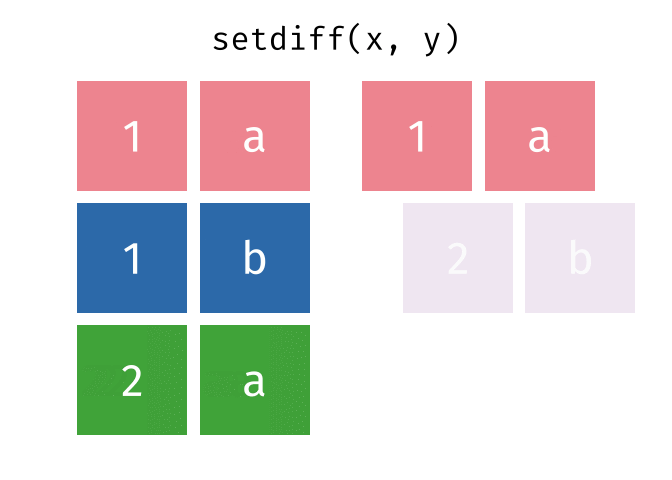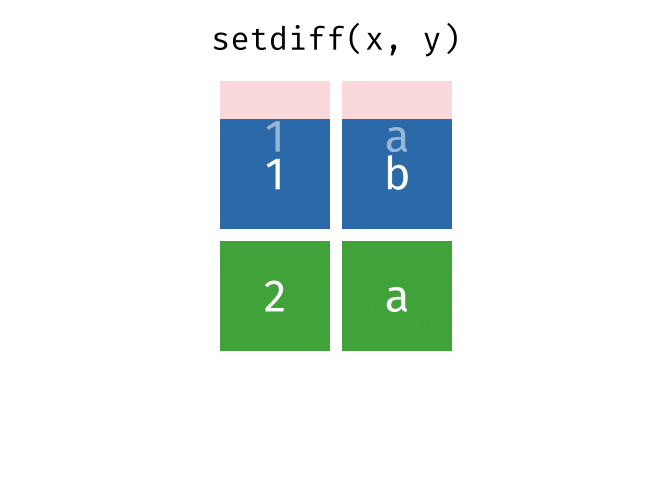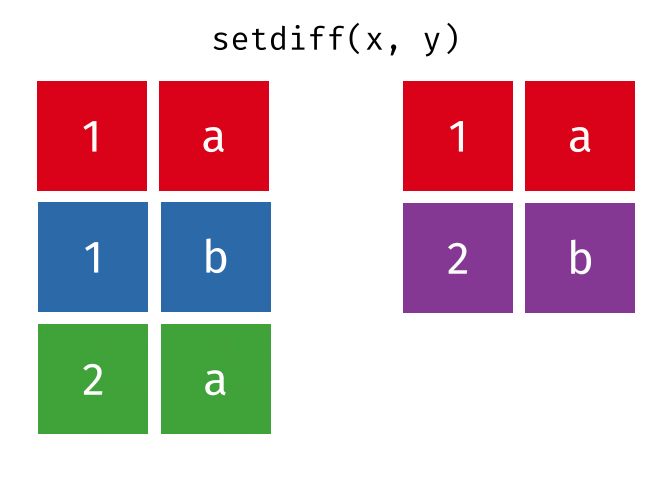
setdiff()
setdiff() gibt die Zeilen zurück, die in der ersten, aber nicht in der zweiten Tabelle vorkommen - also die Differenzmenge.
setdiff(menge_a, menge_b)# A tibble: 1 × 2
stadt land
<chr> <chr>
1 Berlin DeutschlandBerlin ist nur in menge_a.
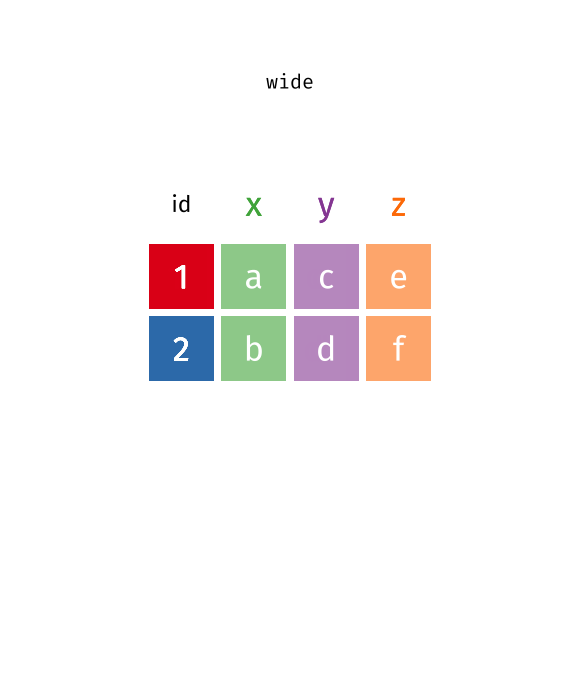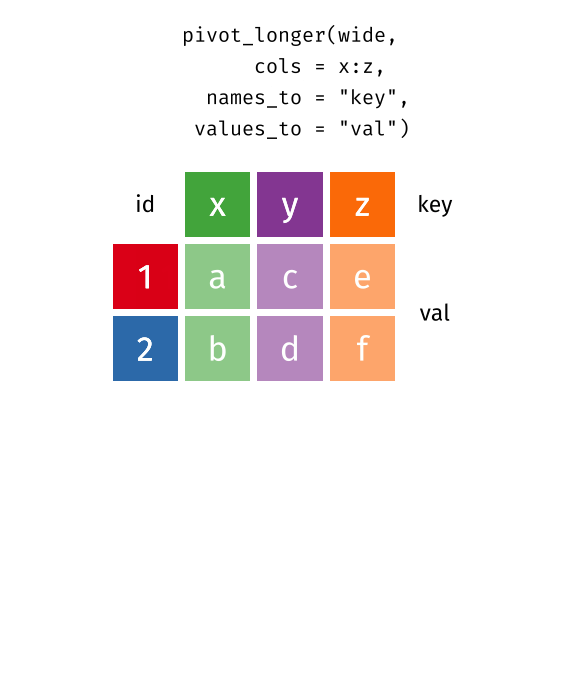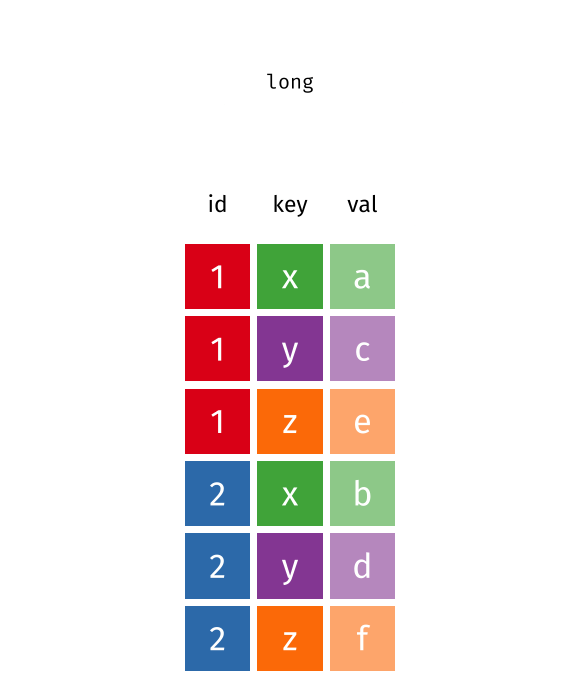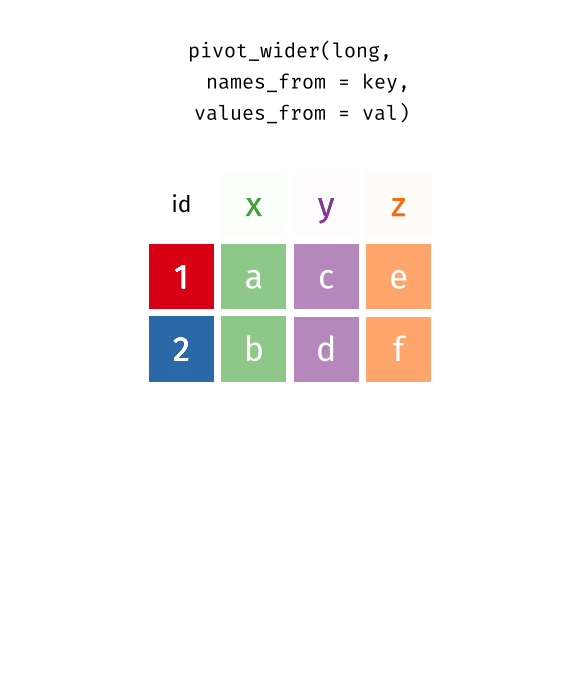
Daten umstrukturieren (Wide ↔︎ Long)
Oft muss man Daten zwischen zwei Formaten transformieren:
- Wide Format (breit): Jede Variable hat eine eigene Spalte
- Long Format (lang): Variablennamen werden zu Werten in einer Spalte
Welches Format “richtig” ist, hängt vom Anwendungsfall ab. Für viele tidyverse-Funktionen und ggplot2 ist das Long Format besser geeignet, während das Wide Format oft übersichtlicher für Menschen ist.
pivot_longer()
pivot_longer() transformiert Daten vom Wide ins Long Format - es macht die Tabelle “länger” (mehr Zeilen, weniger Spalten).
Betrachten wir staedte_stats:
staedte_stats# A tibble: 10 × 3
stadt flaeche_km2 gruenflaeche_pct
<chr> <dbl> <dbl>
1 Berlin 892 14.4
2 Hamburg 755 16.8
3 Muenchen 310 11.9
4 Frankfurt 248 21.5
5 Koeln 405 17.2
6 Duesseldorf 217 18.9
7 Stuttgart 207 24
8 Leipzig 297 14.8
9 Dresden 328 12.3
10 Nuernberg 186 19.1Das ist ein typisches Wide Format: Jede Kennzahl (Fläche, Grünfläche) hat eine eigene Spalte. Für manche Analysen oder Visualisierungen möchten wir das in ein Long Format bringen:
staedte_stats %>%
pivot_longer(
cols = c(flaeche_km2, gruenflaeche_pct),
names_to = "kennzahl",
values_to = "wert"
)# A tibble: 20 × 3
stadt kennzahl wert
<chr> <chr> <dbl>
1 Berlin flaeche_km2 892
2 Berlin gruenflaeche_pct 14.4
3 Hamburg flaeche_km2 755
4 Hamburg gruenflaeche_pct 16.8
5 Muenchen flaeche_km2 310
6 Muenchen gruenflaeche_pct 11.9
7 Frankfurt flaeche_km2 248
8 Frankfurt gruenflaeche_pct 21.5
9 Koeln flaeche_km2 405
10 Koeln gruenflaeche_pct 17.2
11 Duesseldorf flaeche_km2 217
12 Duesseldorf gruenflaeche_pct 18.9
13 Stuttgart flaeche_km2 207
14 Stuttgart gruenflaeche_pct 24
15 Leipzig flaeche_km2 297
16 Leipzig gruenflaeche_pct 14.8
17 Dresden flaeche_km2 328
18 Dresden gruenflaeche_pct 12.3
19 Nuernberg flaeche_km2 186
20 Nuernberg gruenflaeche_pct 19.1Die wichtigsten Argumente:
-
cols: Welche Spalten sollen “zusammengeklappt” werden? -
names_to: Wie soll die neue Spalte heißen, die die alten Spaltennamen enthält? -
values_to: Wie soll die neue Spalte heißen, die die Werte enthält?
Jetzt hat jede Stadt zwei Zeilen - eine pro Kennzahl. Das ist ideal für ggplot2, wenn man z.B. beide Kennzahlen in einem Facetten-Plot darstellen möchte.
Spaltenauswahl mit Hilfsfunktionen
Statt die Spalten einzeln aufzulisten, kann man auch Hilfsfunktionen verwenden:
# Alle Spalten außer "stadt"
staedte_stats %>%
pivot_longer(
cols = -stadt,
names_to = "kennzahl",
values_to = "wert"
)# A tibble: 20 × 3
stadt kennzahl wert
<chr> <chr> <dbl>
1 Berlin flaeche_km2 892
2 Berlin gruenflaeche_pct 14.4
3 Hamburg flaeche_km2 755
4 Hamburg gruenflaeche_pct 16.8
5 Muenchen flaeche_km2 310
6 Muenchen gruenflaeche_pct 11.9
7 Frankfurt flaeche_km2 248
8 Frankfurt gruenflaeche_pct 21.5
9 Koeln flaeche_km2 405
10 Koeln gruenflaeche_pct 17.2
11 Duesseldorf flaeche_km2 217
12 Duesseldorf gruenflaeche_pct 18.9
13 Stuttgart flaeche_km2 207
14 Stuttgart gruenflaeche_pct 24
15 Leipzig flaeche_km2 297
16 Leipzig gruenflaeche_pct 14.8
17 Dresden flaeche_km2 328
18 Dresden gruenflaeche_pct 12.3
19 Nuernberg flaeche_km2 186
20 Nuernberg gruenflaeche_pct 19.1# Alle numerischen Spalten
staedte_stats %>%
pivot_longer(
cols = where(is.numeric),
names_to = "kennzahl",
values_to = "wert"
)# A tibble: 20 × 3
stadt kennzahl wert
<chr> <chr> <dbl>
1 Berlin flaeche_km2 892
2 Berlin gruenflaeche_pct 14.4
3 Hamburg flaeche_km2 755
4 Hamburg gruenflaeche_pct 16.8
5 Muenchen flaeche_km2 310
6 Muenchen gruenflaeche_pct 11.9
7 Frankfurt flaeche_km2 248
8 Frankfurt gruenflaeche_pct 21.5
9 Koeln flaeche_km2 405
10 Koeln gruenflaeche_pct 17.2
11 Duesseldorf flaeche_km2 217
12 Duesseldorf gruenflaeche_pct 18.9
13 Stuttgart flaeche_km2 207
14 Stuttgart gruenflaeche_pct 24
15 Leipzig flaeche_km2 297
16 Leipzig gruenflaeche_pct 14.8
17 Dresden flaeche_km2 328
18 Dresden gruenflaeche_pct 12.3
19 Nuernberg flaeche_km2 186
20 Nuernberg gruenflaeche_pct 19.1pivot_wider()
pivot_wider() ist die Umkehrfunktion: Sie transformiert vom Long ins Wide Format - die Tabelle wird “breiter” (weniger Zeilen, mehr Spalten).
Zuerst erstellen wir eine Long-Format-Tabelle:
staedte_long <- staedte_stats %>%
pivot_longer(
cols = -stadt,
names_to = "kennzahl",
values_to = "wert"
)
staedte_long# A tibble: 20 × 3
stadt kennzahl wert
<chr> <chr> <dbl>
1 Berlin flaeche_km2 892
2 Berlin gruenflaeche_pct 14.4
3 Hamburg flaeche_km2 755
4 Hamburg gruenflaeche_pct 16.8
5 Muenchen flaeche_km2 310
6 Muenchen gruenflaeche_pct 11.9
7 Frankfurt flaeche_km2 248
8 Frankfurt gruenflaeche_pct 21.5
9 Koeln flaeche_km2 405
10 Koeln gruenflaeche_pct 17.2
11 Duesseldorf flaeche_km2 217
12 Duesseldorf gruenflaeche_pct 18.9
13 Stuttgart flaeche_km2 207
14 Stuttgart gruenflaeche_pct 24
15 Leipzig flaeche_km2 297
16 Leipzig gruenflaeche_pct 14.8
17 Dresden flaeche_km2 328
18 Dresden gruenflaeche_pct 12.3
19 Nuernberg flaeche_km2 186
20 Nuernberg gruenflaeche_pct 19.1Jetzt transformieren wir zurück ins Wide Format:
staedte_long %>%
pivot_wider(
names_from = kennzahl,
values_from = wert
)# A tibble: 10 × 3
stadt flaeche_km2 gruenflaeche_pct
<chr> <dbl> <dbl>
1 Berlin 892 14.4
2 Hamburg 755 16.8
3 Muenchen 310 11.9
4 Frankfurt 248 21.5
5 Koeln 405 17.2
6 Duesseldorf 217 18.9
7 Stuttgart 207 24
8 Leipzig 297 14.8
9 Dresden 328 12.3
10 Nuernberg 186 19.1Die wichtigsten Argumente:
-
names_from: Welche Spalte enthält die zukünftigen Spaltennamen? -
values_from: Welche Spalte enthält die Werte?
Möglicherweise hast du in diesem Kontext bereits andere Funktionen verwendet. Hier sind einige Alternativen, die mittlerweile teilweise veraltet sind:
Typischer Anwendungsfall: Kreuztabellen
pivot_wider() ist auch nützlich, um Kreuztabellen zu erstellen. Angenommen, wir haben Verkaufsdaten:
# A tibble: 4 × 3
produkt quartal umsatz
<chr> <chr> <dbl>
1 Apfel Q1 100
2 Apfel Q2 120
3 Birne Q1 80
4 Birne Q2 90verkaeufe %>%
pivot_wider(
names_from = quartal,
values_from = umsatz
)# A tibble: 2 × 3
produkt Q1 Q2
<chr> <dbl> <dbl>
1 Apfel 100 120
2 Birne 80 90Jetzt haben wir eine übersichtliche Kreuztabelle mit Produkten in den Zeilen und Quartalen in den Spalten.
Übung: Pivoting-Workflow
Bereite zunächst einen Datensatz im Long-Format vor:
# PlantGrowth mit mehreren Messungen simulieren
set.seed(42)
pflanzen_long <- PlantGrowth %>%
mutate(
plant_id = 1:n(),
hoehe_cm = weight * 5 + rnorm(n(), mean = 0, sd = 2)
) %>%
pivot_longer(
cols = c(weight, hoehe_cm),
names_to = "messung",
values_to = "wert"
) %>%
select(plant_id, group, messung, wert)
pflanzen_long# A tibble: 60 × 4
plant_id group messung wert
<int> <fct> <chr> <dbl>
1 1 ctrl weight 4.17
2 1 ctrl hoehe_cm 23.6
3 2 ctrl weight 5.58
4 2 ctrl hoehe_cm 26.8
5 3 ctrl weight 5.18
6 3 ctrl hoehe_cm 26.6
7 4 ctrl weight 6.11
8 4 ctrl hoehe_cm 31.8
9 5 ctrl weight 4.5
10 5 ctrl hoehe_cm 23.3
# ℹ 50 more rowsFühre die folgenden Transformationen durch:
Transformiere
pflanzen_longins Wide-Format, sodassweightundhoehe_cmjeweils eigene Spalten sind.Füge eine neue Spalte
bmi(Body Mass Index für Pflanzen) hinzu, die das Verhältnisweight / hoehe_cmberechnet.Transformiere den Datensatz zurück ins Long-Format, sodass nun alle drei Variablen (
weight,hoehe_cmundbmi) in der Spaltemessungerscheinen.
# a) Wide-Format erstellen
pflanzen_wide <- pflanzen_long %>%
pivot_wider(
names_from = messung,
values_from = wert
)
pflanzen_wide# A tibble: 30 × 4
plant_id group weight hoehe_cm
<int> <fct> <dbl> <dbl>
1 1 ctrl 4.17 23.6
2 2 ctrl 5.58 26.8
3 3 ctrl 5.18 26.6
4 4 ctrl 6.11 31.8
5 5 ctrl 4.5 23.3
6 6 ctrl 4.61 22.8
7 7 ctrl 5.17 28.9
8 8 ctrl 4.53 22.5
9 9 ctrl 5.33 30.7
10 10 ctrl 5.14 25.6
# ℹ 20 more rows# A tibble: 30 × 5
plant_id group weight hoehe_cm bmi
<int> <fct> <dbl> <dbl> <dbl>
1 1 ctrl 4.17 23.6 0.177
2 2 ctrl 5.58 26.8 0.208
3 3 ctrl 5.18 26.6 0.195
4 4 ctrl 6.11 31.8 0.192
5 5 ctrl 4.5 23.3 0.193
6 6 ctrl 4.61 22.8 0.202
7 7 ctrl 5.17 28.9 0.179
8 8 ctrl 4.53 22.5 0.202
9 9 ctrl 5.33 30.7 0.174
10 10 ctrl 5.14 25.6 0.201
# ℹ 20 more rows# c) Zurück ins Long-Format (alle drei Variablen)
pflanzen_final_long <- pflanzen_wide %>%
pivot_longer(
cols = c(weight, hoehe_cm, bmi),
names_to = "messung",
values_to = "wert"
)
pflanzen_final_long# A tibble: 90 × 4
plant_id group messung wert
<int> <fct> <chr> <dbl>
1 1 ctrl weight 4.17
2 1 ctrl hoehe_cm 23.6
3 1 ctrl bmi 0.177
4 2 ctrl weight 5.58
5 2 ctrl hoehe_cm 26.8
6 2 ctrl bmi 0.208
7 3 ctrl weight 5.18
8 3 ctrl hoehe_cm 26.6
9 3 ctrl bmi 0.195
10 4 ctrl weight 6.11
# ℹ 80 more rowsZusammenfassung
Gut gemacht! Man beherrscht jetzt die wichtigsten Techniken, um Tabellen in R zu kombinieren und umzustrukturieren.
-
Tabellen stapeln:
-
bind_rows(): Zeilen untereinander stapeln - funktioniert auch bei unterschiedlichen Spalten (fehlende werden mit NA gefüllt) -
bind_cols(): Spalten nebeneinander kleben - Vorsicht: keine intelligente Verknüpfung, Reihenfolge muss stimmen!
-
-
Mutating Joins (fügen Spalten hinzu):
-
left_join(): Alle Zeilen aus der linken Tabelle behalten - der Standardfall -
right_join(): Alle Zeilen aus der rechten Tabelle behalten -
inner_join(): Nur Zeilen mit Partner in beiden Tabellen -
full_join(): Alle Zeilen aus beiden Tabellen
-
-
Filtering Joins (nur filtern, keine neuen Spalten):
-
semi_join(): Zeilen aus x, die einen Partner in y haben -
anti_join(): Zeilen aus x, die keinen Partner in y haben - ideal für “Was fehlt?”-Fragen
-
-
Set Operations (Tabellen als Mengen, brauchen identische Spalten):
-
union(): Alle einzigartigen Zeilen aus beiden -
intersect(): Nur Zeilen, die in beiden vorkommen -
setdiff(): Zeilen aus x, die nicht in y sind
-
-
Pivoting (Datenformat ändern):
-
pivot_longer(): Wide → Long (mehr Zeilen, weniger Spalten) -
pivot_wider(): Long → Wide (weniger Zeilen, mehr Spalten)
-
-
Best Practices:
- Bei unterschiedlichen Spaltennamen:
by = c("name_links" = "name_rechts") - Im Zweifel
left_join()stattbind_cols() -
anti_join()zur Datenqualitätsprüfung nutzen
- Bei unterschiedlichen Spaltennamen:
Zitat
@online{schmidt2026,
author = {{Dr. Paul Schmidt}},
publisher = {BioMath GmbH},
title = {1. Tabellen kombinieren},
date = {2026-02-07},
url = {https://biomathcontent.netlify.app/de/content/r_more/01_joins.html},
langid = {de}
}