---
title: "8. Eigene Funktionen"
subtitle: "Wiederverwendbaren Code schreiben und tidyeval verstehen"
---
```{r}
#| label: func-setup
#| include: false
# Packages
for (pkg in c("tidyverse", "rlang")) {
if (!require(pkg, character.only = TRUE)) install.packages(pkg)
}
library(tidyverse)
library(rlang)
```
## Warum eigene Funktionen?
Einer der wichtigsten Schritte auf dem Weg vom R-Anwender zum R-Programmierer ist das Schreiben eigener Funktionen. Das Grundprinzip dahinter ist einfach: Wenn man denselben Code mehr als zweimal kopiert und eingefügt hat, sollte man eine Funktion daraus machen. Diese Regel wird oft als **DRY-Prinzip** bezeichnet — "Don't Repeat Yourself".
Das Schreiben von Funktionen hat mehrere handfeste Vorteile. Erstens kann man der Funktion einen aussagekräftigen Namen geben, der sofort verrät, was der Code tut. Zweitens muss man bei Änderungen nur eine einzige Stelle im Code anpassen, nicht jede Kopie. Drittens eliminiert man Fehler, die beim Kopieren und Einfügen entstehen — etwa wenn man vergisst, einen Variablennamen an einer Stelle zu ändern. Und viertens kann man Funktionen projektübergreifend wiederverwenden.
Betrachten wir folgenden Code, der Spalten auf einen Wertebereich von 0 bis 1 skaliert:
```{r}
#| label: func-motivation-problem
mtcars %>%
select(mpg, hp, wt, qsec) %>%
mutate(
mpg = (mpg - min(mpg, na.rm = TRUE)) / (max(mpg, na.rm = TRUE) - min(mpg, na.rm = TRUE)),
hp = (hp - min(hp, na.rm = TRUE)) / (max(hp, na.rm = TRUE) - min(hp, na.rm = TRUE)),
wt = (wt - min(wt, na.rm = TRUE)) / (max(hp, na.rm = TRUE) - min(wt, na.rm = TRUE)),
qsec = (qsec - min(qsec, na.rm = TRUE)) / (max(qsec, na.rm = TRUE) - min(qsec, na.rm = TRUE))
) %>%
head()
```
Dieser Code ist nicht nur lang und repetitiv, er enthält auch einen subtilen Fehler — lässt er sich finden? Bei so viel Wiederholung ist es fast unvermeidlich, dass sich Tippfehler einschleichen. Eine Funktion löst beide Probleme.
:::{.callout-tip}
## Weiterführende Ressourcen
Dieses Kapitel orientiert sich stark am hervorragenden [Kapitel 25: Functions](https://r4ds.hadley.nz/functions.html) aus "R for Data Science" (2. Auflage). Für eine noch tiefere Behandlung von Tidy Evaluation empfehlen wir die [Programming with dplyr](https://dplyr.tidyverse.org/articles/programming.html) Vignette.
:::
## Grundlagen: function()
### Syntax und Aufbau
Eine Funktion in R besteht aus drei Teilen: einem **Namen**, den **Argumenten** und dem **Body** (Funktionskörper). Die grundlegende Syntax sieht so aus:
```{r}
#| label: func-basic-syntax
#| eval: false
funktionsname <- function(argument1, argument2) {
# Body: Der Code, der ausgeführt wird
ergebnis <- argument1 + argument2
ergebnis
}
```
Wenden wir das auf unser Skalierungsproblem an. Der sich wiederholende Teil ist die Formel `(x - min(x)) / (max(x) - min(x))`. Das einzige, was sich ändert, ist die Variable — das wird unser Argument:
```{r}
#| label: func-rescale01
rescale01 <- function(x) {
rng <- range(x, na.rm = TRUE)
(x - rng[1]) / (rng[2] - rng[1])
}
```
Testen wir die Funktion:
```{r}
#| label: func-rescale01-test
rescale01(c(-10, 0, 10))
rescale01(c(1, 2, 3, NA, 5))
```
Jetzt wird unser ursprünglicher Code deutlich lesbarer und kürzer:
```{r}
#| label: func-rescale01-usage
mtcars %>%
select(mpg, hp, wt, qsec) %>%
mutate(
mpg = rescale01(mpg),
hp = rescale01(hp),
wt = rescale01(wt),
qsec = rescale01(qsec)
) %>%
head()
```
### Argumente mit und ohne Defaults
Funktionen können beliebig viele Argumente haben. Argumente ohne Default-Wert sind **erforderlich**, Argumente mit Default-Wert sind **optional**:
```{r}
#| label: func-arguments-demo
# na.rm hat einen Default-Wert, x nicht
my_mean <- function(x, na.rm = FALSE) {
sum(x, na.rm = na.rm) / length(x)
}
my_mean(c(1, 2, 3))
my_mean(c(1, 2, NA), na.rm = TRUE)
```
Bei der Reihenfolge gilt: Erforderliche Argumente zuerst, optionale danach. Das wichtigste Argument (meist die Daten) kommt an erste Stelle — das ermöglicht die nahtlose Integration in Pipe-Ketten.
### Rueckgabewerte
R-Funktionen geben automatisch das Ergebnis der letzten Zeile zurück. Man kann auch explizit `return()` verwenden, was besonders bei frühem Abbruch nützlich ist:
```{r}
#| label: func-return-demo
# Implizite Rückgabe (letzte Zeile)
add_one <- function(x) {
x + 1
}
# Explizite Rückgabe mit return()
safe_divide <- function(x, y) {
if (y == 0) {
return(NA_real_)
}
x / y
}
safe_divide(10, 2)
safe_divide(10, 0)
```
Die Konvention ist: `return()` nur für frühe Abbrüche verwenden. Am Ende der Funktion ist die implizite Rückgabe üblicher und lesbarer.
### Das Ellipsis-Argument (...)
Das spezielle Argument `...` (drei Punkte, auch "Ellipsis" genannt) erlaubt es, beliebig viele zusätzliche Argumente an eine andere Funktion durchzureichen:
```{r}
#| label: func-ellipsis-demo
# Alle zusätzlichen Argumente werden an mean() weitergegeben
my_summary <- function(x, ...) {
c(
mean = mean(x, ...),
sd = sd(x, ...)
)
}
# Ohne na.rm
my_summary(c(1, 2, 3))
# Mit na.rm = TRUE (wird an mean() und sd() durchgereicht)
my_summary(c(1, 2, NA), na.rm = TRUE)
```
Das ist besonders nützlich, wenn man Wrapper-Funktionen schreibt und nicht alle möglichen Argumente der inneren Funktion explizit auflisten möchte.
:::{.callout-tip collapse="false"}
## Übung: Standardabweichung vom Mittelwert
Schreibe eine Funktion `cv()`, die den Variationskoeffizienten (Standardabweichung geteilt durch Mittelwert) berechnet. Die Funktion sollte ein optionales `na.rm`-Argument haben.
```{r}
#| label: func-exercise-cv-task
#| eval: false
cv(c(1, 2, 3, 4, 5))
cv(c(1, 2, NA, 4, 5), na.rm = TRUE)
```
:::
:::{.callout-note collapse="true"}
## Lösung
```{r}
#| label: func-exercise-cv-solution
cv <- function(x, na.rm = FALSE) {
sd(x, na.rm = na.rm) / mean(x, na.rm = na.rm)
}
cv(c(1, 2, 3, 4, 5))
cv(c(1, 2, NA, 4, 5), na.rm = TRUE)
```
:::
## Drei Funktionstypen
Das R for Data Science Buch unterscheidet drei nützliche Kategorien von Funktionen, die man häufig schreibt.
### Vektor-Funktionen
Vektor-Funktionen nehmen einen oder mehrere Vektoren als Input und geben einen Vektor zurück. Sie lassen sich weiter unterteilen in **Mutate-Funktionen** (Output hat gleiche Länge wie Input) und **Summary-Funktionen** (Output hat Länge 1).
```{r}
#| label: func-vector-types
# Mutate-Funktion: gleiche Länge wie Input
z_score <- function(x) {
(x - mean(x, na.rm = TRUE)) / sd(x, na.rm = TRUE)
}
z_score(c(1, 2, 3, 4, 5))
# Summary-Funktion: ein Wert als Output
coef_variation <- function(x, na.rm = FALSE) {
sd(x, na.rm = na.rm) / mean(x, na.rm = na.rm)
}
coef_variation(c(1, 2, 3, 4, 5))
```
### Dataframe-Funktionen
Dataframe-Funktionen nehmen einen Dataframe als Input und geben einen Dataframe zurück. Sie sind typischerweise Wrapper um dplyr-Verben:
```{r}
#| label: func-dataframe-type
# Beispiel einer Dataframe-Funktion
filter_extreme <- function(df, var, threshold = 2) {
df %>%
filter(abs(as.vector(scale({{ var }}))) > threshold)
}
# Autos mit extremem Verbrauch (> 2 SD vom Mittelwert)
mtcars %>%
filter_extreme(mpg) %>%
select(mpg, hp, wt)
```
### Plot-Funktionen
Plot-Funktionen nehmen einen Dataframe und geben einen ggplot zurück:
```{r}
#| label: func-plot-type
#| fig-width: 5
#| fig-height: 3
# Beispiel einer Plot-Funktion
histogram <- function(df, var, binwidth = NULL) {
df %>%
ggplot(aes(x = {{ var }})) +
geom_histogram(binwidth = binwidth)
}
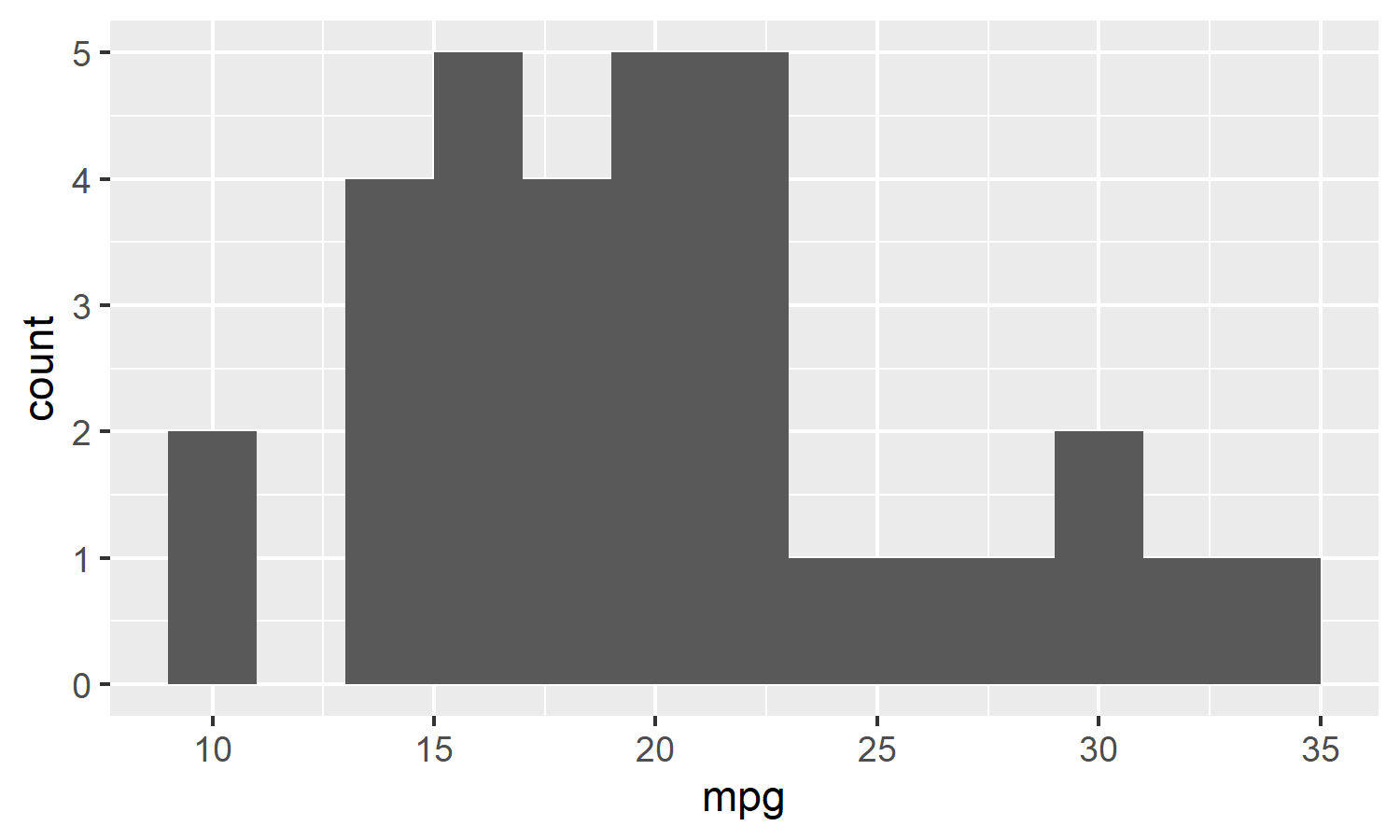
mtcars %>% histogram(mpg, binwidth = 2)
```
Die `{ }`-Syntax im Plot-Beispiel wird im Abschnitt über Tidy Evaluation genauer erklärt.
## Defensive Programmierung
Gute Funktionen prüfen ihre Eingaben und geben verständliche Fehlermeldungen aus. Das spart Debugging-Zeit und macht den Code robuster.
### stop() fuer Fehlermeldungen
Die Funktion `stop()` bricht die Ausführung ab und zeigt eine Fehlermeldung an:
```{r}
#| label: func-stop-demo
#| error: true
calculate_bmi <- function(weight_kg, height_m) {
if (!is.numeric(weight_kg) || !is.numeric(height_m)) {
stop("weight_kg und height_m muessen numerisch sein")
}
if (any(height_m <= 0)) {
stop("height_m muss positiv sein")
}
weight_kg / height_m^2
}
calculate_bmi(70, 1.75)
calculate_bmi(70, "groß")
```
### stopifnot() fuer schnelle Checks
Für einfache Bedingungen ist `stopifnot()` kompakter:
```{r}
#| label: func-stopifnot-demo
#| error: true
calculate_bmi <- function(weight_kg, height_m) {
stopifnot(is.numeric(weight_kg), is.numeric(height_m))
stopifnot(all(height_m > 0))
weight_kg / height_m^2
}
calculate_bmi(70, 0)
```
Der Nachteil: Die automatisch generierten Fehlermeldungen sind weniger informativ als selbst formulierte.
### match.arg() fuer kategorische Argumente
Wenn ein Argument nur bestimmte Werte annehmen darf, verwendet man `match.arg()`:
```{r}
#| label: func-matcharg-demo
#| error: true
center <- function(x, type = c("mean", "median", "trimmed")) {
type <- match.arg(type)
switch(type,
mean = mean(x, na.rm = TRUE),
median = median(x, na.rm = TRUE),
trimmed = mean(x, trim = 0.1, na.rm = TRUE)
)
}
center(1:10, "mean")
center(1:10, "median")
center(1:10, "mena")
```
Die erlaubten Werte werden im Default des Arguments definiert. `match.arg()` erlaubt auch partielle Übereinstimmung und gibt hilfreiche Fehlermeldungen bei falschen Eingaben.
:::{.callout-tip collapse="false"}
## Übung: Sichere Logarithmus-Funktion
Schreibe eine Funktion `safe_log()`, die:
1. Prüft, ob der Input numerisch ist
2. Prüft, ob alle Werte positiv sind
3. Bei negativen Werten eine hilfreiche Fehlermeldung gibt, die anzeigt, wie viele nicht-positive Werte vorhanden sind
```{r}
#| label: func-exercise-safelog-task
#| eval: false
safe_log(c(1, 10, 100))
safe_log(c(-1, 10, 100))
```
:::
:::{.callout-note collapse="true"}
## Lösung
```{r}
#| label: func-exercise-safelog-solution
#| error: true
safe_log <- function(x, base = exp(1)) {
if (!is.numeric(x)) {
stop("x muss numerisch sein, nicht ", typeof(x))
}
n_negative <- sum(x <= 0, na.rm = TRUE)
if (n_negative > 0) {
stop(
glue::glue("x enthält {n_negative} Wert(e) <= 0. ",
"Logarithmus ist nur für positive Zahlen definiert.")
)
}
log(x, base = base)
}
safe_log(c(1, 10, 100))
safe_log(c(-1, 0, 10, 100))
```
:::
## Funktionen im tidyverse: Tidy Evaluation
Sobald man Funktionen schreibt, die tidyverse-Verben wie `filter()`, `mutate()` oder `ggplot()` verwenden, stößt man auf ein besonderes Problem: Wie übergibt man Spaltennamen als Argumente?
### Das Problem: Indirection
Betrachten wir diese naive Funktion:
```{r}
#| label: func-indirection-problem
#| error: true
grouped_mean <- function(df, group_var, mean_var) {
df %>%
group_by(group_var) %>%
summarize(mean = mean(mean_var))
}
mtcars %>% grouped_mean(cyl, mpg)
```
Die Funktion sucht nach Spalten namens `group_var` und `mean_var` — aber die gibt es nicht! Das Problem ist **Indirection**: dplyr verwendet **Data Masking**, um Spaltennamen ohne Anführungszeichen zu erlauben. Das ist praktisch im interaktiven Gebrauch, aber macht das Schreiben von Funktionen komplizierter.
:::{.callout-note}
## Data Masking erklärt
Data Masking bedeutet, dass man `filter(df, x > 5)` schreiben kann statt `filter(df, df$x > 5)`. R sucht `x` zuerst in den Spalten des Dataframes, dann in der Umgebung. Das ist der Grund, warum `group_var` als Spaltenname interpretiert wird — nicht als Variable, die einen Spaltennamen enthält.
:::
### Die Standardloesung: Curly-Curly
Seit rlang 0.4.0 (2019) gibt es eine elegante Lösung: den **Embracing-Operator** `{ }` (auch "curly-curly" genannt). Er sagt dplyr: "Schau nicht nach einer Spalte mit diesem Namen, sondern schau in diese Variable hinein":
```{r}
#| label: func-curly-curly-solution
grouped_mean <- function(df, group_var, mean_var) {
df %>%
group_by({{ group_var }}) %>%
summarize(mean = mean({{ mean_var }}), .groups = "drop")
}
mtcars %>% grouped_mean(cyl, mpg)
```
Die Regel ist einfach: **Jedes Argument, das an eine tidyverse-Funktion weitergegeben wird, die Data Masking oder Tidy Selection verwendet, muss embraced werden.**
Woher weiß man, welche Funktionen das sind? Die Dokumentation verrät es: Man sucht nach `<data-masking>` (für Funktionen wie `filter()`, `mutate()`, `summarize()`) oder `<tidy-select>` (für Funktionen wie `select()`, `rename()`, `across()`).
```{r}
#| label: func-curly-curly-examples
# Flexible Summary-Funktion
summary_stats <- function(df, var) {
df %>%
summarize(
n = n(),
mean = mean({{ var }}, na.rm = TRUE),
sd = sd({{ var }}, na.rm = TRUE),
min = min({{ var }}, na.rm = TRUE),
max = max({{ var }}, na.rm = TRUE)
)
}
mtcars %>% summary_stats(mpg)
mtcars %>% group_by(cyl) %>% summary_stats(mpg)
```
:::{.callout-tip collapse="false"}
## Übung: Proportionen zählen
Schreibe eine Funktion `count_prop()`, die wie `count()` funktioniert, aber zusätzlich eine Spalte `prop` mit dem Anteil hinzufügt.
```{r}
#| label: func-exercise-countprop-task
#| eval: false
# Gewünschtes Ergebnis:
mtcars %>% count_prop(cyl)
# # A tibble: 3 × 3
# cyl n prop
# <dbl> <int> <dbl>
# 1 4 11 0.344
# 2 6 7 0.219
# 3 8 14 0.438
```
:::
:::{.callout-note collapse="true"}
## Lösung
```{r}
#| label: func-exercise-countprop-solution
count_prop <- function(df, var, sort = FALSE) {
df %>%
count({{ var }}, sort = sort) %>%
mutate(prop = n / sum(n))
}
mtcars %>% count_prop(cyl)
```
:::
### Dynamische Spaltennamen mit dem Walrus-Operator
Was, wenn man nicht nur eine Spalte *lesen*, sondern eine Spalte mit dynamischem Namen *erstellen* möchte? Der normale `=`-Operator erlaubt links nur feste Namen. Hier kommt `:=` ins Spiel (der "Walrus-Operator"):
```{r}
#| label: func-walrus-operator
# Funktion, die eine neue Spalte mit dynamischem Namen erstellt
standardize <- function(df, var) {
df %>%
mutate(
"{{ var }}_z" := ({{ var }} - mean({{ var }}, na.rm = TRUE)) /
sd({{ var }}, na.rm = TRUE)
)
}
mtcars %>%
select(mpg, cyl) %>%
standardize(mpg) %>%
head()
```
Die Syntax `"{{ var }}_z" :=` kombiniert die glue-artige String-Interpolation mit dem Walrus-Operator. Das `{ var }` im String wird durch den Variablennamen ersetzt.
### Spalten als Strings: .data Pronoun
Manchmal hat man Spaltennamen als Strings — etwa aus einer Konfigurationsdatei oder Benutzereingabe. Hier verwendet man das `.data`-Pronoun:
```{r}
#| label: func-data-pronoun
# Spaltenname kommt als String
summarize_column <- function(df, col_name) {
df %>%
summarize(mean = mean(.data[[col_name]], na.rm = TRUE))
}
summarize_column(mtcars, "mpg")
# Nützlich für Iteration über Spaltennamen
col_names <- c("mpg", "hp", "wt")
map(col_names, ~ summarize_column(mtcars, .x))
```
### Fortgeschritten: enquo() und !!
Die `{ }`-Syntax ist eine Kurzschreibweise für eine Kombination aus `enquo()` und `!!`. In den meisten Fällen braucht man die explizite Form nicht, aber es gibt Situationen, wo sie nötig ist — zum Beispiel wenn man den Variablennamen als String extrahieren möchte.
Hier dieselbe Funktion in beiden Schreibweisen:
```{r}
#| label: func-enquo-comparison
# Mit {{ }} - die empfohlene Kurzform
grouped_mean_short <- function(df, group_var, mean_var) {
df %>%
group_by({{ group_var }}) %>%
summarize(mean = mean({{ mean_var }}), .groups = "drop")
}
# Mit enquo() und !! - die explizite Form
grouped_mean_explicit <- function(df, group_var, mean_var) {
group_var <- enquo(group_var) # Argument einfangen
mean_var <- enquo(mean_var)
df %>%
group_by(!!group_var) %>% # Mit !! wieder einsetzen
summarize(mean = mean(!!mean_var), .groups = "drop")
}
# Beide liefern dasselbe Ergebnis
mtcars %>% grouped_mean_short(cyl, mpg)
mtcars %>% grouped_mean_explicit(cyl, mpg)
```
**enquo()** fängt ein Argument ein, ohne es auszuwerten. **!!** (bang-bang) fügt den eingefangenen Ausdruck wieder ein.
Wann braucht man die explizite Form? Wenn man den Variablennamen als String extrahieren möchte:
```{r}
#| label: func-enquo-aslabel
# as_label() extrahiert den Namen als String - nur mit enquo() möglich
summary_with_label <- function(df, var) {
var_quo <- enquo(var)
var_name <- rlang::as_label(var_quo)
df %>%
summarize(
variable = var_name,
mean = mean(!!var_quo, na.rm = TRUE)
)
}
mtcars %>% summary_with_label(mpg)
mtcars %>% summary_with_label(hp)
```
### Mehrere Spalten als Strings: syms() und !!!
Wenn man mehrere Spaltennamen als Character-Vektor hat und diese in einer tidyverse-Funktion verwenden möchte, braucht man `syms()` und `!!!`:
- **syms()** wandelt einen Character-Vektor in eine Liste von Symbolen um
- **!!!** (splice-Operator) entpackt diese Liste, sodass jedes Element einzeln übergeben wird
```{r}
#| label: func-splice-demo
# Mehrere Gruppierungsvariablen als Character-Vektor
grouped_summary <- function(df, group_vars, summary_var) {
# syms() wandelt c("cyl", "am") in list(sym("cyl"), sym("am")) um
group_symbols <- syms(group_vars)
df %>%
# !!! entpackt die Liste: group_by(cyl, am) statt group_by(list(...))
group_by(!!!group_symbols) %>%
summarize(mean = mean({{ summary_var }}, na.rm = TRUE), .groups = "drop")
}
mtcars %>% grouped_summary(c("cyl", "am"), mpg)
```
Diese Technik ist besonders nützlich, wenn die Gruppierungsvariablen dynamisch bestimmt werden — etwa aus einer Konfiguration oder Benutzereingabe.
### pick() fuer Tidy Selection in Data-Masking-Kontext
Manchmal möchte man Tidy Selection (wie bei `select()`) innerhalb einer Data-Masking-Funktion (wie `group_by()`) verwenden. Hier hilft `pick()`:
```{r}
#| label: func-pick-demo
# Mehrere Gruppierungsspalten mit Tidy Selection
count_by <- function(df, ...) {
df %>%
group_by(pick(...)) %>%
summarize(n = n(), .groups = "drop")
}
mtcars %>% count_by(cyl, am)
mtcars %>% count_by(starts_with("c"))
```
Wichtig: Bei `...` verwendet man `pick(...)` direkt, nicht `pick({{ ... }})`. Die `{ }`-Syntax ist nur für einzelne benannte Argumente gedacht.
### Uebersicht: Wann welchen Ansatz?
| Situation | Lösung | Beispiel |
|-----------|--------|----------|
| Spalte als "bare name" übergeben | `{ }` | `filter({{ var }} > 0)` |
| Spaltenname als String | `.data[[]]` | `summarize(mean = mean(.data[[col]]))` |
| Mehrere Spalten via `...` | `...` direkt durchreichen | `group_by(...)` oder `pick(...)` |
| Dynamischer Spaltenname erstellen | `:=` | `mutate("{{ var }}_new" := ...)` |
| Variablenname als String extrahieren | `enquo()` + `as_label()` | `as_label(enquo(var))` |
| Liste von Strings zu Symbolen | `syms()` + `!!!` | `group_by(!!!syms(cols))` |
| Tidy Select in Data Masking | `pick()` | `group_by(pick(...))` |
:::{.callout-tip collapse="false"}
## Übung: Flexible Filterung
Schreibe eine Funktion `filter_na()`, die alle Zeilen entfernt, in denen eine bestimmte Spalte `NA` ist.
```{r}
#| label: func-exercise-filtna-task
#| eval: false
# Test-Daten
test_df <- tibble(
x = c(1, NA, 3),
y = c("a", "b", NA)
)
test_df %>% filter_na(x)
test_df %>% filter_na(y)
```
:::
:::{.callout-note collapse="true"}
## Lösung
```{r}
#| label: func-exercise-filtna-solution
filter_na <- function(df, var) {
df %>%
filter(!is.na({{ var }}))
}
test_df <- tibble(
x = c(1, NA, 3),
y = c("a", "b", NA)
)
test_df %>% filter_na(x)
test_df %>% filter_na(y)
```
:::
:::{.callout-tip collapse="false"}
## Übung: Plot-Funktion mit dynamischem Titel
Erweitere die `histogram()`-Funktion so, dass der Titel automatisch den Variablennamen enthält:
```{r}
#| label: func-exercise-plottitle-task
#| eval: false
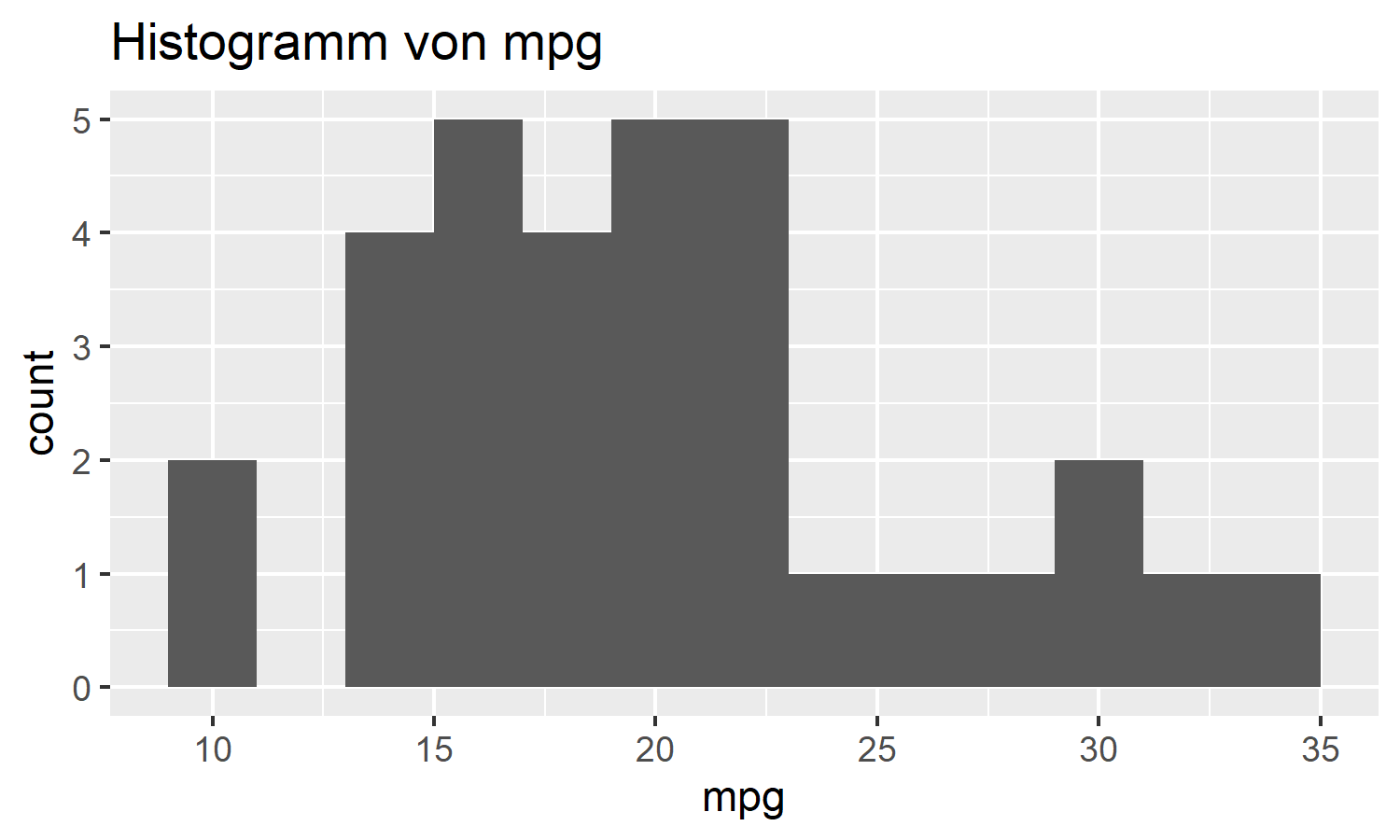
mtcars %>% histogram(mpg, binwidth = 2)
# Sollte einen Titel wie "Histogramm von mpg" haben
```
*Hinweis: `rlang::englue()` oder die Kombination aus `enquo()` und `as_label()` verwenden.*
:::
:::{.callout-note collapse="true"}
## Lösung
```{r}
#| label: func-exercise-plottitle-solution
#| fig-width: 5
#| fig-height: 3
histogram <- function(df, var, binwidth = NULL) {
title <- rlang::englue("Histogramm von {{var}}")
df %>%
ggplot(aes(x = {{ var }})) +
geom_histogram(binwidth = binwidth) +
labs(title = title)
}
mtcars %>% histogram(mpg, binwidth = 2)
```
:::
## Best Practices und Style
### Benennung
Funktionsnamen sollten Verben sein und klar beschreiben, was die Funktion tut:
```{r}
#| label: func-naming
#| eval: false
# Gut: Verben, beschreibend
impute_missing()
calculate_bmi()
extract_coefficients()
# Schlecht: Zu kurz oder nicht beschreibend
f()
my_function()
do_stuff()
```
Argumentnamen sollten Substantive sein. Das Daten-Argument heißt typischerweise `df`, `data` oder `.data`.
### Code-Formatierung
Immer geschweifte Klammern `{}` verwenden, auch bei einzeiligen Funktionen. Der Body wird mit zwei Leerzeichen eingerückt:
```{r}
#| label: func-style
#| eval: false
# Gut
add_one <- function(x) {
x + 1
}
# Vermeiden
add_one <- function(x) x + 1
```
### Dokumentation mit Roxygen
Wenn man ein R-Paket entwickelt, muss jede exportierte Funktion dokumentiert werden. Diese Dokumentation wird im **Roxygen-Format** geschrieben — spezielle Kommentare direkt über der Funktion, die mit `#'` beginnen. Beim Bauen des Pakets werden diese Kommentare automatisch in die formatierten Hilfsseiten umgewandelt, die man mit `?funktionsname` aufruft.
Aber auch wenn man gar kein Paket schreibt, sondern nur ein Skript mit ein paar Hilfsfunktionen, lohnt sich dieses Format. Statt unstrukturierte Kommentare neben die Funktion zu schreiben, kann man gleich das Roxygen-Format verwenden. Es ist übersichtlich, standardisiert, und falls die Funktion später doch in ein Paket wandert, ist die Dokumentation bereits fertig.
Die wichtigsten Roxygen-Tags:
- **Titel** (erste Zeile): Eine kurze, einzeilige Beschreibung der Funktion
- **Beschreibung** (nach Leerzeile): Ausführlichere Erklärung, was die Funktion tut
- **`@param name`**: Beschreibt ein Argument der Funktion
- **`@return`**: Beschreibt, was die Funktion zurückgibt
- **`@examples`**: Ausführbare Beispiele für die Verwendung
```{r}
#| label: func-roxygen-demo
#| eval: false
#' Berechne den Body Mass Index
#'
#' Diese Funktion berechnet den BMI aus Gewicht und Größe.
#' Bei Vektoren wird der BMI elementweise berechnet.
#'
#' @param weight_kg Gewicht in Kilogramm (numerischer Vektor).
#' @param height_m Größe in Metern (numerischer Vektor).
#'
#' @return Ein numerischer Vektor mit BMI-Werten.
#'
#' @examples
#' calculate_bmi(70, 1.75)
#' calculate_bmi(c(60, 80), c(1.60, 1.80))
calculate_bmi <- function(weight_kg, height_m) {
stopifnot(is.numeric(weight_kg), is.numeric(height_m))
stopifnot(all(height_m > 0))
weight_kg / height_m^2
}
```
In RStudio und Positron kann man ein leeres Roxygen-Skelett automatisch einfügen lassen: Cursor in die Funktion setzen und **Code → Insert Roxygen Skeleton** wählen (oder `Ctrl+Alt+Shift+R`).