# Pakete installieren (nur notwendig, falls noch nicht installiert)
for (pkg in c("tidyverse")) {
if (!require(pkg, character.only = TRUE)) install.packages(pkg)
}
# Pakete laden
library(tidyverse)Da wir die absoluten Grundlagen im letzten Abschnitt behandelt haben, sind wir gespannt darauf, endlich mit echten Tabellen zu arbeiten und nicht nur mit einzelnen Werten und Vektoren. Und genau das werden wir jetzt tun. Dies ist jedoch auch ein guter Punkt, um über das Tidyverse zu sprechen.
Das Tidyverse ist nicht nur ein einzelnes, sondern eine Sammlung mehrerer R-Pakete, die zusammenarbeiten und - einfach ausgedrückt - die Verwendung von R für alle Arten der Datenverarbeitung gleichzeitig einfacher, schneller und mächtiger machen. Es folgen mehrere Vergleiche zwischen der Durchführung von Aufgaben mit base R einerseits und mit dem Tidyverse andererseits. Man muss verstehen, dass R all diese Dinge auch ohne die Verwendung von Funktionen und Paketen aus dem Tidyverse bewältigen kann - schließlich existierte R lange Zeit ohne die Tidyverse-Pakete. Das Tidyverse ist jedoch ein sehr populärer und mächtiger Weg, Dinge zu erledigen, und ich bin nicht der einzige, der es der base R-Methode vorzieht.
Um alle in diesem Kapitel verwendeten Pakete zu installieren und zu laden, führt man folgenden Code aus:
Man bemerkt, dass wir eine ziemlich lange Ausgabe erhalten, wenn wir das Paket namens Tidyverse laden. Das liegt u.a. daran, dass das Tidyverse nicht nur ein Paket ist, sondern eine Sammlung mehrerer Pakete. Der library(tidyverse)-Befehl lädt sie alle auf einmal. Das ist eine sehr praktische Funktion, da wir normalerweise mehrere Pakete aus dem Tidyverse gleichzeitig verwenden. Daher listet der erste Teil der Ausgabe einfach alle 9 Pakete auf, die geladen wurden. Der zweite Teil über die Konflikte wird später besprochen.
Wie man oben sehen kann, habe ich ein # nicht nur zum Schreiben von Kommentaren verwendet, sondern auch zum “Auskommentieren” von Code. Genauer gesagt habe ich ein # vor den install.packages("tidyverse")-Befehl gesetzt, was bedeutet, dass dieser Befehl nicht ausgeführt wird, wenn man den Code ausführt, aber der Code ist leicht verfügbar, falls man ihn ausführen muss.
Das ist eine sehr nützliche Funktion in R und vielen anderen Programmiersprachen. Wenn man einen Befehl ausführen möchte, aber nicht gerade jetzt, kann man ihn auskommentieren und den Rest des Codes ausführen. Man beachte, dass es sogar eine Tastenkombination in RStudio zum Auskommentieren von Code gibt: Nachdem man eine oder sogar mehrere Zeilen Code markiert hat, drückt man Ctrl + Shift + C, um den Code auszukommentieren. Wenn man dieselbe Kombination erneut drückt, wird der Code wieder einkommentiert.
Tabellen
Tabelle in base R: data.frame
Für uns sind Tabellen wahrscheinlich die wichtigste Datenstruktur in R. Oft sind die Daten in einer .xlsx-, .csv- oder ähnlichen Datei gespeichert und man liest sie dann in R ein. Eine Tabelle in R wird in der base R-Terminologie als data.frame bezeichnet. Hier ist ein Beispiel, wie man selbst eine erstellt und in einer Variable namens my_df speichert:
(In der Praxis erstellt man offensichtlich nicht oft Tabellen manuell, wie unten gezeigt. Wir werden bald das Importieren von Daten besprechen.)
my_df <- data.frame(
name = c("Wei", "Priya", "Kwame", "Juan"),
age = c(25, 30, 35, 28),
height = c(180, 170, 190, 175)
)
my_df name age height
1 Wei 25 180
2 Priya 30 170
3 Kwame 35 190
4 Juan 28 175Wie man sehen kann, haben wir eine Funktion namens data.frame() verwendet, um eine Tabelle mit drei Spalten zu erstellen: name, age und height. Diese drei funktionieren also nicht als vordefinierte Argumentnamen wie bei der seq()-Funktion im letzten Abschnitt, sondern stattdessen als Namen der zu erstellenden Spalten. Außerdem wird der Inhalt jeder Spalte durch einen Vektor der Länge 4 definiert, sodass letztendlich eine Tabelle mit 3 Spalten und 4 Zeilen erstellt wird.
Man beachte, dass ein data.frame wirklich eine Sammlung von Vektoren ist. Diese Tatsache hilft dabei zu verstehen, wie man mit Tabellen in R arbeitet. Zur Erinnerung: Ein Vektor ist eine Sammlung von Werten desselben Typs. Im obigen Beispiel ist die name-Spalte ein Vektor von character-Werten, die age-Spalte ist ein Vektor von numerischen Werten und die height-Spalte ist ebenfalls ein Vektor von numerischen Werten. Man kann versuchen, über Daten nachzudenken, mit denen man im täglichen Leben arbeitet, und es ist wahrscheinlich, dass jede Spalte Werte desselben Typs enthält.
Man hat mehrere Möglichkeiten, auf Teile eines data.frame zuzugreifen. Zum Beispiel kann man auf eine einzelne Spalte zugreifen, indem man den $-Operator verwendet. Dadurch wird der einzelne Vektor zurückgegeben, der die Spalte repräsentiert:
my_df$name[1] "Wei" "Priya" "Kwame" "Juan" Man kann auch die eckigen Klammern ([]) verwenden, um bestimmte Teile der Tabelle zu extrahieren - genau wie wir es mit Vektoren im letzten Abschnitt getan haben. Man muss jedoch daran denken, dass eine Tabelle zweidimensional ist und daher sowohl die Zeile als auch die Spalte angeben muss, auf die man zugreifen möchte. Das kann man mit der [Zeile, Spalte]-Syntax tun. Um zum Beispiel auf das Alter der zweiten Person in der Tabelle zuzugreifen, kann man verwenden:
my_df[2, "age"] # alternativ: my_df[2, 2] da age die zweite Spalte ist[1] 30Schließlich gibt es hier zwei Funktionen, die man typischerweise in jedem R-Tutorial findet, um die Struktur einer Tabelle zu untersuchen:
str(my_df)'data.frame': 4 obs. of 3 variables:
$ name : chr "Wei" "Priya" "Kwame" "Juan"
$ age : num 25 30 35 28
$ height: num 180 170 190 175Zuerst sehen wir, dass der data.frame 4 Beobachtungen (=Zeilen) von 3 Variablen (=Spalten) hat. Dann erhalten wir eine Art Übersicht über jede Spalte, die uns ihre Namen, Datentypen und die ersten paar Werte anzeigt (in diesem Fall alle Werte).
summary(my_df) name age height
Length:4 Min. :25.00 Min. :170.0
Class :character 1st Qu.:27.25 1st Qu.:173.8
Mode :character Median :29.00 Median :177.5
Mean :29.50 Mean :178.8
3rd Qu.:31.25 3rd Qu.:182.5
Max. :35.00 Max. :190.0 Diese Funktion liefert auch Informationen über die Spalten der Tabelle, aber auf eine andere Weise. Zum Beispiel: für numerische Spalten gibt sie das Minimum, 1. Quartil, Median, Mittelwert, 3. Quartil und den Maximalwert an.
Tabelle im Tidyverse: tibble
Alles, was man bisher gesehen hat, ist die base R-Art, mit Tabellen umzugehen. Nun schauen wir uns an, wie das Tidyverse es macht. Das Tidyverse hat seine eigene Tabellenstruktur namens tibble. Laut den Autoren ist das tibble “eine moderne Neuvorstellung des data.frame, die beibehält, was sich als effektiv erwiesen hat, und das verwirft, was es nicht ist.” Mit anderen Worten, das tibble ist etwas benutzerfreundlicher und hat einige Vorteile gegenüber dem data.frame. Man beachte, dass es keine völlig separate Datenstruktur ist, sondern vielmehr eine modifizierte Version des data.frame - und immer noch auf diesem basiert.
Bevor wir mit tibbles arbeiten können, müssen wir das erforderliche Paket installieren und laden - was wir am Anfang dieses Kapitels getan haben. Wenn man nach oben zur Liste der Pakete scrollt, die beim Laden des Tidyverse angezeigt wurde, wird man bemerken, dass eines davon tibble hieß. Das ist das Paket, das die tibble-Datenstruktur und alle dazugehörigen Funktionen bereitstellt.
Hier ist, wie wir dieselbe Tabelle wie oben erstellen würden, aber diesmal als tibble und in einer Variable namens my_tbl speichern:
my_tbl <- tibble(
name = c("Wei", "Priya", "Kwame", "Juan"),
age = c(25, 30, 35, 28),
height = c(180, 170, 190, 175)
)
my_tbl# A tibble: 4 × 3
name age height
<chr> <dbl> <dbl>
1 Wei 25 180
2 Priya 30 170
3 Kwame 35 190
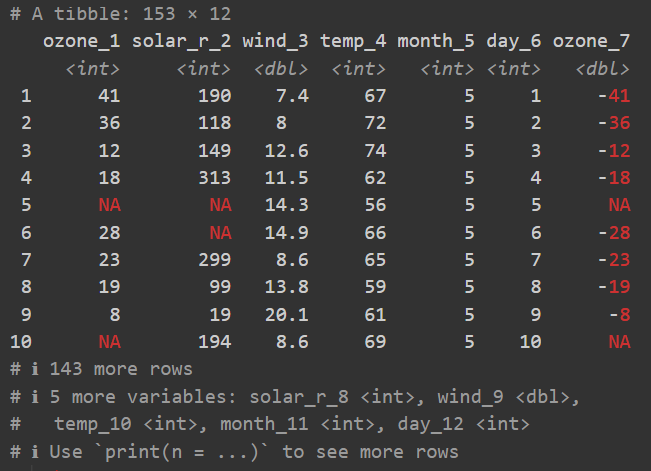
4 Juan 28 175Man beachte, dass es bezüglich des Codes wirklich nur einen Unterschied zur base R-Art der Tabellenerstellung gibt: Wir verwenden die tibble()-Funktion anstelle der data.frame()-Funktion. Die Ausgabe ist jedoch etwas anders und hier kommen die Vorteile des tibble zum Tragen. Obwohl es immer noch dieselbe Tabelle in Bezug auf ihren Inhalt ist, wird das tibble automatisch auf eine benutzerfreundlichere Weise ausgegeben. Um möglichst alle Vorteile zu zeigen, wird hier beispielhaft ein größerer Datensatz ausgegeben. Dieser hat 153 Zeilen, 12 Spalten, Fehlwerte (NA) und negative Werte:
Man sieht also:
- Es gibt eine zusätzliche erste Zeile, die uns über die Anzahl der Zeilen und Spalten informiert.
- Es gibt eine zusätzliche Zeile unter den Spaltennamen, die uns über den Datentyp jeder Spalte informiert.
- Nur die ersten zehn Datenzeilen werden ausgegeben.
- Nur die ersten Spalten werden ausgegeben.
- Fehlende Werte
NAund negative Zahlen werden rot ausgegeben.
All diese kleinen Dinge summieren sich wirklich über die Zeit und machen das Arbeiten mit tibbles angenehmer als mit data.frames. Schließlich ist zu beachten, dass ein tibble in seinem Kern immer noch ein data.frame ist und man in den meisten Fällen alles mit einem tibble machen kann, was man mit einem data.frame machen kann. Hier sind dieselben Befehle, die wir oben verwendet haben, als Beweis:
my_tbl$name[1] "Wei" "Priya" "Kwame" "Juan" str(my_tbl)tibble [4 × 3] (S3: tbl_df/tbl/data.frame)
$ name : chr [1:4] "Wei" "Priya" "Kwame" "Juan"
$ age : num [1:4] 25 30 35 28
$ height: num [1:4] 180 170 190 175summary(my_tbl) name age height
Length:4 Min. :25.00 Min. :170.0
Class :character 1st Qu.:27.25 1st Qu.:173.8
Mode :character Median :29.00 Median :177.5
Mean :29.50 Mean :178.8
3rd Qu.:31.25 3rd Qu.:182.5
Max. :35.00 Max. :190.0 Daher gibt es grundsätzlich keinen Nachteil und stattdessen nur Vorteile bei der Verwendung von tibbles gegenüber data.frames. Und was hier für Tabellen gilt, veranschaulicht auch die allgemeine Idee des Tidyverse: Es ist nicht unbedingt eine völlig neue Art, Dinge zu tun, sondern vielmehr eine benutzerfreundlichere und mächtigere Art, Dinge zu tun, die bereits vorher möglich waren.
An dieser Stelle möchte ich ein kostenloses Online-Buch empfehlen, das eine großartige Ressource zum Erlernen des Tidyverse und R im Allgemeinen ist: “R for Data Science (2e)”. Es wurde von Hadley Wickham, Mine Çetinkaya-Rundel und Garrett Grolemund geschrieben, die selbst Autoren einiger der wichtigsten Tidyverse-Pakete sind.
Ein neuer Beispieldatensatz
Bevor wir weitermachen, verwerfen wir die kleinen Tabellen, die wir oben erstellt haben, und verwenden stattdessen einen Datensatz, der mit R geliefert wird. Dieser Datensatz heißt PlantGrowth und enthält Daten zum Gewicht von 30 Pflanzen in 3 verschiedenen Gruppen. Da er sozusagen in R eingebaut ist (genau wie pi; siehe letztes Kapitel), können wir direkt über seinen Namen darauf zugreifen. Es ist jedoch ein data.frame und wir möchten stattdessen mit einem tibble arbeiten. Daher konvertieren wir es mit der as_tibble()-Funktion in ein tibble und speichern es in einer Variable namens tbl:
tbl <- as_tibble(PlantGrowth)
tbl# A tibble: 30 × 2
weight group
<dbl> <fct>
1 4.17 ctrl
2 5.58 ctrl
3 5.18 ctrl
4 6.11 ctrl
5 4.5 ctrl
6 4.61 ctrl
7 5.17 ctrl
8 4.53 ctrl
9 5.33 ctrl
10 5.14 ctrl
# ℹ 20 more rowsWir sehen, dass nicht alle 30 Datenzeilen, sondern stattdessen nur die ersten 10 Zeilen anzeigt werden und darunter ein “… with 20 more rows” Hinweis steht. Das ist eine sehr nützliche Funktion beim Arbeiten mit großen Datensätzen.
Diagramme
Diagramm in base R: plot()
Base R hat eine plot()-Funktion, die gut darin ist, erste Datenvisualisierungen mit sehr wenig Code zu erstellen. Sie errät, welche Art von Diagramm man sehen möchte, über den Datentyp der jeweiligen zu plottenden Daten:
Diagramm im Tidyverse: ggplot()
Ich verwende plot() jedoch wirklich nur, um einen schnellen ersten Blick auf Daten zu werfen. Um professionelle Visualisierungen zu erhalten, verwende ich immer das Tidyverse-Paket {ggplot2} und seine Funktion ggplot(). Es scheint, als könnte es jedes vorstellbare Diagramm erstellen. Seine hohe Leistungsfähigkeit geht jedoch mit dem Preis einer langen Lernkurve einher. Daher verweise ich vorerst nur auf zusätzliche Ressourcen und werde in den nächsten Abschnitten einige Grundlagen behandeln. Als Appetitanreger ist hier jedoch ein Screenshot einiger mit ggplot erstellter Diagramme von Cédric Scherer:
Der Pipe-Operator
Der Pipe-Operator (%>% oder |>) “veränderte völlig die Art, wie wir in R programmieren, und machte es einfacher und lesbarer” (Álvarez, 2001). Wir haben angefangen, die Pipe als %>% aus dem {dplyr}-Paket zu verwenden1. Seit dem 18. Mai 2021 (= R 4.1.0) ist die Pipe jedoch offiziell Teil von Base R - obwohl als |> geschrieben. Man beachte, dass es einige Unterschiede zwischen %>% und |> gibt - mehr dazu findet man z.B. hier, hier oder hier.
Um zu verstehen, was ihn so großartig macht, müssen wir anfangen, mehr als eine Funktion gleichzeitig zu verwenden. Bisher haben wir Funktionen nur einzeln verwendet. Im wirklichen Leben wird man jedoch oft feststellen, dass man mehrere Funktionen kombinieren muss.
Als Beispiel nehmen wir an, wir haben drei Zahlen 1, 4 und 10 (d.h. einen Vektor c(1, 4, 10)) und wir möchten (i) ihre Quadratwurzel nehmen, dann (ii) den Mittelwert dieser Werte ermitteln und (iii) die Quadratwurzel dieses Mittelwerts nehmen. Schließlich möchten wir das Ergebnis in einer Variable namens result speichern.
Bevor du weiterliest: Versuche dies mit dem bereits vorhandenen Wissen zu erreichen, d.h. ohne den Pipe-Operator.
Siehe die folgenden drei Abschnitte für verschiedene Lösungsansätze.
Lösung 1: Zwischenergebnisse
Eine Möglichkeit, unser Ziel hier zu erreichen, ist es schrittweise zu tun und jedes Zwischenergebnis in einer Variable zu speichern. Das ist ein sehr häufiger Ansatz in der Programmierung und wird “schrittweiser” oder “iterativer” Ansatz genannt. So würde es in R aussehen:
x <- c(1, 4, 10)
step1 <- sqrt(x) # Schritt 1: Quadratwurzel nehmen
step2 <- mean(step1) # Schritt 2: Mittelwert ermitteln
result <- sqrt(step2) # Schritt 3: Quadratwurzel des Mittelwerts nehmen
result[1] 1.433211Das funktioniert perfekt und es ist leicht zu lesen, da wir jeden Schritt sehen können. Es braucht jedoch auch ziemlich viel Code und erstellt Variablen, die uns nicht wirklich interessieren.
Lösung 2: Verschachtelte Funktionen
Eine andere Möglichkeit, dasselbe Ergebnis zu erreichen, ist die Verwendung verschachtelter Funktionen - genau wie man es in Microsoft Excel tun würde. Das bedeutet, dass wir eine Funktion in eine andere Funktion setzen:
x <- c(1, 4, 10)
result <- sqrt(mean(sqrt(x)))
result[1] 1.433211Der offensichtliche Vorteil ist, dass es weniger Code braucht und keine unnötigen Variablen erstellt. Es ist jedoch auch weniger lesbar, weil man von innen nach außen lesen muss. Das ist bei einfachen Funktionen wie dieser kein Problem, aber es kann bei komplexeren Funktionen sehr verwirrend werden.
Lösung 3: Der Pipe-Operator
Dies kombiniert die Vorteile beider obigen Ansätze, da er (i) es ermöglicht, Funktionen von links nach rechts / oben nach unten zu schreiben und somit in der Reihenfolge, in der sie ausgeführt werden und wie man über sie denkt, und (ii) keine zusätzlichen Variablen für Zwischenschritte erstellt:
x <- c(1, 4, 10)
result <- x %>% sqrt() %>% mean() %>% sqrt()
result[1] 1.433211Man kann es sich so vorstellen: Etwas (in diesem Fall x) geht in die Pipe und wird zur nächsten Funktion sqrt() geleitet. Standardmäßig nimmt diese Funktion das, was aus der vorherigen Pipe herauskam, und setzt es als ihr erstes Argument ein. Das passiert bei jeder Pipe.
Vielleicht hilft es zu erkennen, dass diese beiden identische Dinge tun:
sqrt(9)9 %>% sqrt()
Die Tastenkombination zum Schreiben von %>% in RStudio ist CTRL+SHIFT+M. Tastenkombinationen können in RStudio angepasst werden, wie hier beschrieben.
dplyr-Verben
Wir haben nun eine Vorstellung davon, wie das Erstellen von Tabellen, Diagrammen und generell das Programmieren schöner ist, wenn man das Tidyverse verwendet. Ein weiterer sehr wichtiger Teil des Tidyverse ist das Paket {dplyr}. Dieses Paket stellt eine Reihe von Funktionen bereit, die sehr nützlich für die Datenmanipulation sind. Diese Funktionen werden oft als “Verben” bezeichnet, weil sie beschreiben, was man mit seinen Daten machen möchte. Direkt aus der Dokumentation übernommen:
{dplyr} ist eine Grammatik der Datenmanipulation, die eine konsistente Reihe von Verben bereitstellt, die dabei helfen, die häufigsten Herausforderungen der Datenmanipulation zu lösen:
mutate()fügt neue Variablen hinzu, die Funktionen bestehender Variablen sind.
select()wählt Variablen basierend auf ihren Namen aus.
filter()wählt Fälle basierend auf ihren Werten aus.
summarise()reduziert mehrere Werte auf eine einzige Zusammenfassung.
arrange()ändert die Reihenfolge der Zeilen.Diese alle kombinieren sich natürlich mit
group_by(), was es ermöglicht, jede Operation “nach Gruppen” durchzuführen. Wenn man neu bei dplyr ist, ist der beste Startpunkt das Kapitel zur Datentransformation in R for data science.
Nach unserer Erfahrung kann man wirklich den größten Teil der Datenmanipulation vor und nach der eigentlichen Statistik mit diesen Funktionen erledigen. Mit anderen Worten, es sind genau diese Funktionen, die die manuelle Arbeit ersetzen können und sollten, die man möglicherweise gerade in MS Excel macht, um seine Daten zu bearbeiten. In den folgenden Abschnitten geben wir sehr kurze Beispiele, wie diese Funktionen zu verwenden sind, während wir immer auf gründlichere Ressourcen verweisen.
Bevor wir anfangen, sie zu verwenden, erstellen wir einige Spielzeugdatensätze, mit denen es schön zu arbeiten ist. Bitte ignoriert, dass wir einige der unten verwendeten Funktionen noch nicht eingeführt haben. Wir werden sie in den nächsten Abschnitten behandeln. Vorerst wollen wir nur diese vier Datensätze erstellen, mit denen wir arbeiten können:
dat1 <- as_tibble(PlantGrowth)
dat2 <- dat1 %>% head() # erste 6 Zeilen behalten
dat3 <- dat1 %>% slice(1:4, 11:14, 21:24) # Zeilen 1-4, 11-14 und 21-24 behalten
dat4 <- dat1 %>% slice(1, 2, 11, 12, 21, 22) %>% # Zeilen 1, 2, 11, 12, 21 und 22 behalten
mutate(var1 = 1:6, var2 = 22:27, var3 = 3:8, var4 = 4:9)Hier ist etwas, was man verstehen muss, bevor wir weitermachen.
Was man oben sieht, ist Code, der 4 neue Datensätze/Objekte/Variablen (dat1 - dat3) erstellt hat, jeder durch das Nehmen eines anderen Datensatzes und dessen Manipulation auf irgendeine Weise. Der erste Datensatz ist der eingebaute Datensatz PlantGrowth, aber als tibble formatiert. Der Grund, warum die manipulierte Version (d.h. die tibble-Version von PlantGrowth) dauerhaft als dat1 verfügbar ist, liegt daran, dass wir den <--Operator verwendet und diese manipulierte Version in dieser neuen Variable namens dat1 gespeichert haben. Man beachte weiter, dass wir diese neuen Datensätze eigentlich nicht sehen. Wenn wir dat1 sehen wollten, müssten wir dat1 (oder print(dat1)) ausführen, damit sein Inhalt in die Konsole ausgegeben wird.
In den folgenden Abschnitten werden wir sehr viele Datenmanipulationen durchführen, weil das ist, was wir lernen werden. Wir werden jedoch eigentlich nie die manipulierten Datensätze in neuen Variablen speichern. Stattdessen werden wir den Code ohne das ... <- ausführen und somit immer einfach die manipulierte Version des Datensatzes in die Konsole ausgeben. Das ist gut für den Zweck zu sehen, was eine Funktion macht. Es ist jedoch offensichtlich nicht das, was man im wirklichen Leben machen wird. Im wirklichen Leben wird man immer die manipulierte Version des Datensatzes in einer neuen Variable speichern.
select()
Die select()-Funktion ermöglicht es, bestimmte Spalten aus einer Tabelle auszuwählen. Das ist sehr nützlich, wenn man eine große Tabelle hat und nur mit wenigen Spalten arbeiten möchte. Das ist also unser Datensatz:
dat4# A tibble: 6 × 6
weight group var1 var2 var3 var4
<dbl> <fct> <int> <int> <int> <int>
1 4.17 ctrl 1 22 3 4
2 5.58 ctrl 2 23 4 5
3 4.81 trt1 3 24 5 6
4 4.17 trt1 4 25 6 7
5 6.31 trt2 5 26 7 8
6 5.12 trt2 6 27 8 9Und wenn ich die select()-Funktion verwende, um die Spalte group auszuwählen, wird sie eine neue Tabelle mit nur dieser Spalte zurückgeben:
dat4 %>% select(group)# A tibble: 6 × 1
group
<fct>
1 ctrl
2 ctrl
3 trt1
4 trt1
5 trt2
6 trt2 Außerdem kann man mehr als eine Spalte benennen:
dat4 %>% select(group, var2, var4)# A tibble: 6 × 3
group var2 var4
<fct> <int> <int>
1 ctrl 22 4
2 ctrl 23 5
3 trt1 24 6
4 trt1 25 7
5 trt2 26 8
6 trt2 27 9Nochmals eine Erinnerung, dass der Pipe-Operator (%>%) hier nicht unbedingt notwendig ist. Die Funktion select() funktioniert für sich allein und sie benötigt die Daten als ihr erstes Argument. Daher könnte man auch select(dat4, group) oder select(dat4, group, var2, var4) schreiben. Wir werden jedoch weiterhin den Pipe-Operator verwenden, weil er den Code leichter zu lesen und zu verstehen macht - zumindest auf lange Sicht.
Man kann sogar mehrere Spalten auf einmal auswählen, indem man den :-Operator verwendet. Wenn man zum Beispiel alle Spalten von var2 bis var4 auswählen möchte, kann man das so machen:
dat4 %>% select(group, var2:var4)# A tibble: 6 × 4
group var2 var3 var4
<fct> <int> <int> <int>
1 ctrl 22 3 4
2 ctrl 23 4 5
3 trt1 24 5 6
4 trt1 25 6 7
5 trt2 26 7 8
6 trt2 27 8 9Man kann auch den --Operator verwenden, um bestimmte Spalten auszuschließen. Wenn man zum Beispiel alle Spalten außer var1 auswählen möchte, kann man das so machen:
dat4 %>% select(-var1)# A tibble: 6 × 5
weight group var2 var3 var4
<dbl> <fct> <int> <int> <int>
1 4.17 ctrl 22 3 4
2 5.58 ctrl 23 4 5
3 4.81 trt1 24 5 6
4 4.17 trt1 25 6 7
5 6.31 trt2 26 7 8
6 5.12 trt2 27 8 9Schließlich gibt es mehrere Hilfsfunktionen, die es ermöglichen, Spalten basierend auf ihren Namen auszuwählen. Wenn man zum Beispiel alle Spalten auswählen möchte, die mit “var” beginnen, kann man die Hilfsfunktion starts_with() so verwenden:
dat4 %>% select(starts_with("var"))# A tibble: 6 × 4
var1 var2 var3 var4
<int> <int> <int> <int>
1 1 22 3 4
2 2 23 4 5
3 3 24 5 6
4 4 25 6 7
5 5 26 7 8
6 6 27 8 9Andere, ähnliche Funktionen sind ends_with(), contains(), matches() und num_range().
Es gibt auch Funktionen wie is.numeric(), is.character() usw., die es ermöglichen, Spalten basierend auf ihrem Datentyp auszuwählen. Wenn man zum Beispiel alle numerischen Spalten auswählen möchte, kann man das so machen:
dat4 %>% select(where(~is.numeric(.x)))# A tibble: 6 × 5
weight var1 var2 var3 var4
<dbl> <int> <int> <int> <int>
1 4.17 1 22 3 4
2 5.58 2 23 4 5
3 4.81 3 24 5 6
4 4.17 4 25 6 7
5 6.31 5 26 7 8
6 5.12 6 27 8 9Das sind sehr mächtige Funktionen der select()-Funktion und ermöglichen es, Spalten basierend auf ihren Namen oder Datentypen auszuwählen, ohne sie alle manuell eingeben zu müssen. Schließlich gibt es sogar eine Hilfsfunktion namens everything(), die es ermöglicht, alle Spalten auszuwählen. Das mag zunächst nicht sehr nützlich erscheinen, aber man könnte sie z.B. verwenden, um Spalten neu zu ordnen, indem man spezifische Spalten zuerst auswählt und dann alle anderen Spalten danach:
dat4 %>% select(var2, everything())# A tibble: 6 × 6
var2 weight group var1 var3 var4
<int> <dbl> <fct> <int> <int> <int>
1 22 4.17 ctrl 1 3 4
2 23 5.58 ctrl 2 4 5
3 24 4.81 trt1 3 5 6
4 25 4.17 trt1 4 6 7
5 26 6.31 trt2 5 7 8
6 27 5.12 trt2 6 8 9filter()
Die filter()-Funktion ermöglicht es, Zeilen basierend auf bestimmten Bedingungen zu filtern. Man ist wahrscheinlich damit vertraut aus Excel, wo das auch Filtern genannt wird.
Um etwas zum Filtern zu haben, verwenden wir dat1, da es 30 Beobachtungen hat. Um nur die Beobachtungen zu behalten, bei denen das weight größer als 6 ist, können wir die filter()-Funktion so verwenden:
dat1 %>% filter(weight > 6)# A tibble: 4 × 2
weight group
<dbl> <fct>
1 6.11 ctrl
2 6.03 trt1
3 6.31 trt2
4 6.15 trt2 Man kann eine zweite Bedingung hinzufügen, indem man den &-Operator verwendet, um es so zu machen, dass sowohl Bedingung 1 ALS AUCH Bedingung 2 wahr sein müssen. Wenn man zum Beispiel nur die Beobachtungen behalten möchte, bei denen das weight größer als 6 UND die group “trt2” ist, kann man das so machen:
dat1 %>% filter(weight > 6 & group == "trt2")# A tibble: 2 × 2
weight group
<dbl> <fct>
1 6.31 trt2
2 6.15 trt2 Falls man verwirrt ist, warum wir == anstelle von = schreiben müssen, geht man zurück zum Abschnitt “Vergleichsoperatoren” im vorherigen Kapitel und erinnert sich auch daran, dass ein einzelnes = zum Zuweisen von Werten an Variablen verwendet wird. Hier weisen wir jedoch nichts zu, sondern überprüfen, ob der Wert von group gleich “trt2” ist. Daher müssen wir den doppelten ==-Operator verwenden.
Man kann auch den |-Operator verwenden, um es so zu machen, dass entweder Bedingung 1 ODER Bedingung 2 wahr sein muss. Wir könnten zum Beispiel nur die Beobachtungen behalten, bei denen das weight größer als 6 oder kleiner als 4 ist:
dat1 %>% filter(weight > 6 | weight < 4)# A tibble: 6 × 2
weight group
<dbl> <fct>
1 6.11 ctrl
2 3.59 trt1
3 3.83 trt1
4 6.03 trt1
5 6.31 trt2
6 6.15 trt2 Die nächsten drei Beispiele werden alle zum gleichen Ergebnis führen, aber es auf verschiedene Weise erreichen. Es ist fast immer der Fall, dass es nicht nur einen einzigen Weg gibt, etwas in R zu tun, aber manchmal ist ein Weg effizienter oder leichter zu lesen als ein anderer. Unser Ziel für alle ist es, alle Beobachtungen zu behalten, die nicht zur Kontrollgruppe gehören.
Wir könnten es mit dem |-Operator machen, den wir gerade gelernt haben:
dat1 %>% filter(group == "trt1" | group == "trt2")# A tibble: 20 × 2
weight group
<dbl> <fct>
1 4.81 trt1
2 4.17 trt1
3 4.41 trt1
4 3.59 trt1
5 5.87 trt1
6 3.83 trt1
7 6.03 trt1
8 4.89 trt1
9 4.32 trt1
10 4.69 trt1
11 6.31 trt2
12 5.12 trt2
13 5.54 trt2
14 5.5 trt2
15 5.37 trt2
16 5.29 trt2
17 4.92 trt2
18 6.15 trt2
19 5.8 trt2
20 5.26 trt2 Für Situationen, in denen man mehrere weitere Bedingungen kombinieren müsste, ist der %in%-Operator eine effizientere Möglichkeit, dies zu tun. Er ermöglicht es zu überprüfen, ob ein Wert in einem Vektor von Werten ist. Wir könnten zum Beispiel dasselbe wie oben so machen:
dat1 %>% filter(group %in% c("trt1", "trt2"))# A tibble: 20 × 2
weight group
<dbl> <fct>
1 4.81 trt1
2 4.17 trt1
3 4.41 trt1
4 3.59 trt1
5 5.87 trt1
6 3.83 trt1
7 6.03 trt1
8 4.89 trt1
9 4.32 trt1
10 4.69 trt1
11 6.31 trt2
12 5.12 trt2
13 5.54 trt2
14 5.5 trt2
15 5.37 trt2
16 5.29 trt2
17 4.92 trt2
18 6.15 trt2
19 5.8 trt2
20 5.26 trt2 Schließlich könnten wir auch den !=-Operator verwenden, um zu überprüfen, ob die Gruppe nicht gleich “ctrl” ist:
dat1 %>% filter(group != "ctrl")# A tibble: 20 × 2
weight group
<dbl> <fct>
1 4.81 trt1
2 4.17 trt1
3 4.41 trt1
4 3.59 trt1
5 5.87 trt1
6 3.83 trt1
7 6.03 trt1
8 4.89 trt1
9 4.32 trt1
10 4.69 trt1
11 6.31 trt2
12 5.12 trt2
13 5.54 trt2
14 5.5 trt2
15 5.37 trt2
16 5.29 trt2
17 4.92 trt2
18 6.15 trt2
19 5.8 trt2
20 5.26 trt2 In diesem spezifischen Fall ist die letzte der drei Optionen die kürzeste und am leichtesten zu lesen.
- 5.2 Filter rows with filter() in R for data science
- Subset rows using column values with filter()
arrange()
Die arrange()-Funktion ermöglicht es, die Zeilen einer Tabelle basierend auf den Werten einer oder mehrerer Spalten zu ordnen (d.h. zu sortieren). Hier verwenden wir dat3, das 4 Zeilen für jede der drei Gruppen hat:
dat3# A tibble: 12 × 2
weight group
<dbl> <fct>
1 4.17 ctrl
2 5.58 ctrl
3 5.18 ctrl
4 6.11 ctrl
5 4.81 trt1
6 4.17 trt1
7 4.41 trt1
8 3.59 trt1
9 6.31 trt2
10 5.12 trt2
11 5.54 trt2
12 5.5 trt2 Wir können die Tabelle nach der weight-Spalte so sortieren:
dat3 %>% arrange(weight)# A tibble: 12 × 2
weight group
<dbl> <fct>
1 3.59 trt1
2 4.17 ctrl
3 4.17 trt1
4 4.41 trt1
5 4.81 trt1
6 5.12 trt2
7 5.18 ctrl
8 5.5 trt2
9 5.54 trt2
10 5.58 ctrl
11 6.11 ctrl
12 6.31 trt2 Wie man sehen kann, ist sie standardmäßig aufsteigend sortiert. Wenn man sie absteigend sortieren möchte, kann man die desc()-Hilfsfunktion verwenden und sie um den jeweiligen Spaltennamen wickeln:
dat3 %>% arrange(desc(weight))# A tibble: 12 × 2
weight group
<dbl> <fct>
1 6.31 trt2
2 6.11 ctrl
3 5.58 ctrl
4 5.54 trt2
5 5.5 trt2
6 5.18 ctrl
7 5.12 trt2
8 4.81 trt1
9 4.41 trt1
10 4.17 ctrl
11 4.17 trt1
12 3.59 trt1 Man kann auch nach mehreren Spalten sortieren. Man kann zum Beispiel zuerst nach group und dann nach weight sortieren. Das funktioniert hier wegen der doppelten Werte in der group-Spalte: Die resultierende Tabelle hat die drei Gruppen in alphabetischer Reihenfolge, aber die Zeilen innerhalb jeder Gruppe sind nach weight sortiert:
dat3 %>% arrange(group, weight)# A tibble: 12 × 2
weight group
<dbl> <fct>
1 4.17 ctrl
2 5.18 ctrl
3 5.58 ctrl
4 6.11 ctrl
5 3.59 trt1
6 4.17 trt1
7 4.41 trt1
8 4.81 trt1
9 5.12 trt2
10 5.5 trt2
11 5.54 trt2
12 6.31 trt2 Man beachte, dass man hier group, weight oder beide in die desc()-Funktion einwickeln könnte, wenn man absteigend sortieren wollte.
Schließlich wäre ein etwas fortgeschritteneres Beispiel das Sortieren nach einer bestimmten benutzerdefinierten Reihenfolge. Das ist manchmal notwendig, weil man nicht immer z.B. seine Gruppen in alphabetischer Reihenfolge (oder umgekehrt alphabetischer Reihenfolge) haben möchte. Nehmen wir an, man möchte nach group in der Reihenfolge “trt2”, “ctrl”, “trt1” sortieren. Wir können das erreichen, indem wir unsere benutzerdefinierte Reihenfolge definieren und die Hilfsfunktion match() verwenden:
myorder <- c("trt1", "ctrl", "trt2")
dat3 %>% arrange(match(group, myorder))# A tibble: 12 × 2
weight group
<dbl> <fct>
1 4.81 trt1
2 4.17 trt1
3 4.41 trt1
4 3.59 trt1
5 4.17 ctrl
6 5.58 ctrl
7 5.18 ctrl
8 6.11 ctrl
9 6.31 trt2
10 5.12 trt2
11 5.54 trt2
12 5.5 trt2 Und natürlich könnte man sogar weitergehen und z.B. weight innerhalb jeder Gruppe absteigend sortieren dat3 %>% arrange(match(group, myorder), desc(weight)).
- 5.3 Arrange rows with arrange() in R for data science
- Arrange rows by column values with arrange()
mutate()
Die mutate()-Funktion ermöglicht es, die Werte bestehender Spalten zu mutieren (d.h. zu ändern) oder neue Spalten zu erstellen. Verwenden wir wieder dat2, das nur 6 Zeilen hat, und erstellen eine neue Spalte namens “kg”, die das Gewicht in Kilogramm enthält (d.h. angenommen, weight ist in Gramm, also teilen wir durch 1000):
dat2 %>% mutate(kg = weight / 1000)# A tibble: 6 × 3
weight group kg
<dbl> <fct> <dbl>
1 4.17 ctrl 0.00417
2 5.58 ctrl 0.00558
3 5.18 ctrl 0.00518
4 6.11 ctrl 0.00611
5 4.5 ctrl 0.0045
6 4.61 ctrl 0.00461Wie man sehen kann, funktioniert mutate, indem es (d.h. mit =) einen neuen Spaltennamen (in diesem Fall kg) dem Ergebnis der Operation (in diesem Fall Division durch 1000) auf der bestehenden Spalte weight zuweist.
Wir könnten stattdessen genau dieselbe Operation durchführen, aber sie dem Spaltennamen weight zuweisen. Das wird die bestehende Spalte weight mit den neuen Werten überschreiben oder anders gesagt, es wird die bestehende Spalte weight mutieren/ändern:
dat2 %>% mutate(weight = weight / 1000)# A tibble: 6 × 2
weight group
<dbl> <fct>
1 0.00417 ctrl
2 0.00558 ctrl
3 0.00518 ctrl
4 0.00611 ctrl
5 0.0045 ctrl
6 0.00461 ctrl Wir können auch mehrere Spalten gleichzeitig erstellen und sie müssen nicht mit bestehenden Spalten in Beziehung stehen:
dat2 %>%
mutate(
`Name with Space` = "Hello!",
number10 = 10
)# A tibble: 6 × 4
weight group `Name with Space` number10
<dbl> <fct> <chr> <dbl>
1 4.17 ctrl Hello! 10
2 5.58 ctrl Hello! 10
3 5.18 ctrl Hello! 10
4 6.11 ctrl Hello! 10
5 4.5 ctrl Hello! 10
6 4.61 ctrl Hello! 10Hier werden also zwei Spalten erstellt und einfach mit demselben Wert für alle Zeilen gefüllt. Man beachte, dass der Spaltenname Name with Space Leerzeichen enthält, was in R nicht erlaubt ist. Wenn man es jedoch wirklich will, kann man Backticks (`) verwenden, um Spaltennamen mit Leerzeichen oder anderen Sonderzeichen zu erstellen.
Etwas fortgeschrittener, aber sehr mächtig ist die Kombination von mutate() und case_when(). Das ermöglicht es, neue Spalten basierend auf Bedingungen zu erstellen. Im folgenden Beispiel erstellen wir eine Spalte namens size, die die Werte “large”, “small” oder “normal” enthält, abhängig vom Wert der weight-Spalte. Wenn das Gewicht größer als 5,5 ist, ist es “large”, wenn es kleiner als 4,5 ist, ist es “small” und alles andere ist “normal”:
dat2 %>%
mutate(size = case_when(
weight > 5.5 ~ "large",
weight < 4.5 ~ "small",
TRUE ~ "normal" # alles andere
))# A tibble: 6 × 3
weight group size
<dbl> <fct> <chr>
1 4.17 ctrl small
2 5.58 ctrl large
3 5.18 ctrl normal
4 6.11 ctrl large
5 4.5 ctrl normal
6 4.61 ctrl normalMan kann also sehen, dass die jeweilige Bedingung genauso funktioniert wie bei der filter()-Funktion. Wir schreiben jedoch dann eine Tilde (~) und den Wert, den wir der neuen Spalte zuweisen möchten, wenn die Bedingung wahr ist. Diese Bedingungen werden tatsächlich in der Reihenfolge ausgewertet, in der sie geschrieben sind. Das bedeutet, dass wenn die erste Bedingung wahr ist, die zweite Bedingung nicht ausgewertet wird. Für dieses Beispiel bedeutet das, dass sobald eine Größe auf “large” gesetzt ist, sie nicht auf die folgenden Bedingungen überprüft wird. Wegen diesem Verhalten können wir einfach ein TRUE als letzte Bedingung setzen, da es einfach für alle verbleibenden Werte wahr sein wird und ihnen den Wert “normal” zuweisen wird.
Man kann so viele Bedingungen haben, wie man möchte, und sie so kompliziert machen, wie man möchte - z.B. mit &- und |-Operatoren. Das kann viel Zeit und manuelle Arbeit sparen.
Schließlich ist eine weitere sehr mächtige Funktionskombination, die viel Zeit und manuelle Arbeit sparen kann, die von mutate() und across(). Sie ist darauf ausgelegt, dabei zu helfen, Änderungen an mehreren Spalten gleichzeitig vorzunehmen. Vielleicht muss man zum Beispiel nicht nur die weight-Spalte in Kilogramm umwandeln, sondern auch die var1-, var2-, var3- und var4-Spalten. Sicher, man könnte das ohne across() so machen:
dat4 %>%
mutate(
weight = weight / 1000,
var1 = var1 / 1000,
var2 = var2 / 1000,
var3 = var3 / 1000,
var4 = var4 / 1000
)# A tibble: 6 × 6
weight group var1 var2 var3 var4
<dbl> <fct> <dbl> <dbl> <dbl> <dbl>
1 0.00417 ctrl 0.001 0.022 0.003 0.004
2 0.00558 ctrl 0.002 0.023 0.004 0.005
3 0.00481 trt1 0.003 0.024 0.005 0.006
4 0.00417 trt1 0.004 0.025 0.006 0.007
5 0.00631 trt2 0.005 0.026 0.007 0.008
6 0.00512 trt2 0.006 0.027 0.008 0.009Man stelle sich jedoch vor, man hätte 500 statt 5 Spalten zu bearbeiten. Es ist viel effizienter, die across()-Funktion zu verwenden. Hier ist, wie es funktioniert:
dat4 %>% mutate(across(c(weight, var1:var4), ~ .x / 1000))# A tibble: 6 × 6
weight group var1 var2 var3 var4
<dbl> <fct> <dbl> <dbl> <dbl> <dbl>
1 0.00417 ctrl 0.001 0.022 0.003 0.004
2 0.00558 ctrl 0.002 0.023 0.004 0.005
3 0.00481 trt1 0.003 0.024 0.005 0.006
4 0.00417 trt1 0.004 0.025 0.006 0.007
5 0.00631 trt2 0.005 0.026 0.007 0.008
6 0.00512 trt2 0.006 0.027 0.008 0.009Ja, das sieht ganz anders aus als die Art, wie wir mutate() bis hier verwendet haben, aber es ist immer dieselbe Struktur:
mutate(across(TEIL1, TEIL2))- TEIL1: Die Spalten, die man mutieren möchte.
- TEIL2: Die Operation, die man auf diesen Spalten durchführen möchte - mit
.xals Platzhalter für die Spaltenwerte.
Das Auswählen der Spalten in TEIL1 funktioniert genauso wie bei der select()-Funktion, also kann man dieselben Hilfsfunktionen wie starts_with(), ends_with(), contains(), where(is.numeric()) usw. verwenden. TEIL2 erwartet eine Funktion und in unserem Fall brauchen wir den ~-Operator, um R zu sagen, dass es eine Funktion erstellen soll, die die Eingabe .x nimmt und durch 1000 teilt.
Verwende dat1 und schreibe eine einzelne Pipe, die folgende Schritte umsetzt (nicht unbedingt in dieser Reihenfolge):
- Behalte nur die Zeilen, in denen
weightgrößer als 5 ist - Füge eine neue Spalte namens
weight_kghinzu, die das Gewicht geteilt durch 1000 enthält - Sortiere das Ergebnis absteigend nach
weight - Behalte nur die Spalten
groupundweight_kg
Das Endergebnis sollte ein Tibble mit 2 Spalten und weniger als 30 Zeilen sein.
dat1 %>%
filter(weight > 5) %>%
mutate(weight_kg = weight / 1000) %>%
arrange(desc(weight)) %>%
select(group, weight_kg)# A tibble: 17 × 2
group weight_kg
<fct> <dbl>
1 trt2 0.00631
2 trt2 0.00615
3 ctrl 0.00611
4 trt1 0.00603
5 trt1 0.00587
6 trt2 0.0058
7 ctrl 0.00558
8 trt2 0.00554
9 trt2 0.0055
10 trt2 0.00537
11 ctrl 0.00533
12 trt2 0.00529
13 trt2 0.00526
14 ctrl 0.00518
15 ctrl 0.00517
16 ctrl 0.00514
17 trt2 0.00512Hinweis: Die Reihenfolge von filter(), mutate() und arrange() könnte geändert werden, ohne das Ergebnis zu beeinflussen. Allerdings muss select() am Ende stehen (oder zumindest nach mutate() und filter()), da wir die weight-Spalte für diese Operationen benötigen.
summarize()
Die summarize()-Funktion ermöglicht es, eine Tabelle zusammenzufassen, indem man zusammenfassende Statistiken für eine oder mehrere Spalten berechnet.
Wir verwenden wieder dat1, das 30 Zeilen hat. Nehmen wir an, wir möchten das mittlere Gewicht aller Pflanzen im Datensatz berechnen. Das können wir mit der summarize()-Funktion so machen:
dat1 %>% summarize(mean_weight = mean(weight))# A tibble: 1 × 1
mean_weight
<dbl>
1 5.07Das wird eine neue Tabelle mit einer einzigen Spalte namens mean_weight zurückgeben, die das mittlere Gewicht aller Pflanzen im Datensatz enthält. Man beachte, dass die Syntax ziemlich ähnlich zu der von mutate() ist, aber anstatt eine neue Spalte zur bestehenden Tabelle hinzuzufügen, erstellt sie eine neue Tabelle mit den zusammenfassenden Statistiken.
Bisher ist das eigentlich nicht sehr nützlich, da wir auch einfach das hier hätten machen können: mean(dat1$weight), um diese Zahl zu erhalten. Die wahre Macht von summarize() kommt jedoch ins Spiel, wenn man zusammenfassende Statistiken für mehrere Gruppen berechnen möchte und summarize() und die group_by()-Funktion so kombiniert:
dat1 %>%
group_by(group) %>%
summarize(mean_weight = mean(weight))# A tibble: 3 × 2
group mean_weight
<fct> <dbl>
1 ctrl 5.03
2 trt1 4.66
3 trt2 5.53Wie man sehen kann, erhalten wir sofort das mittlere Gewicht für jede Gruppe. Das liegt daran, dass die group_by()-Funktion grundsätzlich den Daten sagt, alle folgenden Funktionen auf jede Gruppe separat anzuwenden. In diesem Fall sagt sie der summarize()-Funktion, das mittlere Gewicht für jede Gruppe separat zu berechnen. Das kann also viel Zeit und manuelle Arbeit sparen, wenn man viele Gruppen hat.
Es wird noch besser, wenn man alle anderen deskriptiven Statistiken hinzufügt, die man berechnen möchte. Wenn man zum Beispiel den Mittelwert, die Standardabweichung, den Median, das Minimum und das Maximum des Gewichts für jede Gruppe berechnen möchte, kann man das so machen:
dat1 %>%
group_by(group) %>%
summarize(
mean_weight = mean(weight),
median_weight = median(weight),
sd_weight = sd(weight),
min_weight = min(weight),
max_weight = max(weight)
)# A tibble: 3 × 6
group mean_weight median_weight sd_weight min_weight max_weight
<fct> <dbl> <dbl> <dbl> <dbl> <dbl>
1 ctrl 5.03 5.15 0.583 4.17 6.11
2 trt1 4.66 4.55 0.794 3.59 6.03
3 trt2 5.53 5.44 0.443 4.92 6.31Man kann also grundsätzlich die gesamte deskriptive Statistiktabelle in einem Zug erstellen.
Und nur um sicherzustellen, dass das klar ist: Gruppierung muss nicht nur für eine einzelne Variable sein. Man kann sehr wohl ein Experiment mit mehreren Faktoren haben und möchte das mittlere Gewicht für jede Kombination dieser Faktoren berechnen. In diesem Fall kann man einfach mehr Variablen zur group_by()-Funktion hinzufügen. Wir können einen solchen zweiten Faktor zu dat3 so hinzufügen:
dat3 %>%
mutate(factor2 = rep(x = c("A", "B"), times = 6))# A tibble: 12 × 3
weight group factor2
<dbl> <fct> <chr>
1 4.17 ctrl A
2 5.58 ctrl B
3 5.18 ctrl A
4 6.11 ctrl B
5 4.81 trt1 A
6 4.17 trt1 B
7 4.41 trt1 A
8 3.59 trt1 B
9 6.31 trt2 A
10 5.12 trt2 B
11 5.54 trt2 A
12 5.5 trt2 B Und dann in der group_by()-Funktion verwenden:
dat3 %>%
mutate(factor2 = rep(x = c("A", "B"), times = 6)) %>%
group_by(group, factor2) %>%
summarize(mean_weight = mean(weight))`summarise()` has grouped output by 'group'. You can override using the
`.groups` argument.# A tibble: 6 × 3
# Groups: group [3]
group factor2 mean_weight
<fct> <chr> <dbl>
1 ctrl A 4.68
2 ctrl B 5.85
3 trt1 A 4.61
4 trt1 B 3.88
5 trt2 A 5.92
6 trt2 B 5.31Das gibt einem das mittlere Gewicht für jede Kombination von group und factor2.
Schließlich kann man auch die across()-Funktion verwenden, um eine Funktion gleichzeitig auf mehrere Spalten anzuwenden. Wenn man zum Beispiel den Mittelwert pro Gruppe nicht nur für die weight-Spalte, sondern für alle numerischen Spalten in den Daten berechnen möchte, kann man das so machen:
dat4 %>%
group_by(group) %>%
summarize(across(where(is.numeric), ~ mean(.x)))# A tibble: 3 × 6
group weight var1 var2 var3 var4
<fct> <dbl> <dbl> <dbl> <dbl> <dbl>
1 ctrl 4.88 1.5 22.5 3.5 4.5
2 trt1 4.49 3.5 24.5 5.5 6.5
3 trt2 5.72 5.5 26.5 7.5 8.5Und ja, wir können weitergehen und mehr als nur Mittelwerte berechnen. Wenn man zum Beispiel den Mittelwert und die Standardabweichung für alle numerischen Spalten in den Daten berechnen möchte, kann man das so machen:
dat4 %>%
group_by(group) %>%
summarize(across(where(is.numeric), list(mean = ~ mean(.x), sd = ~ sd(.x))))# A tibble: 3 × 11
group weight_mean weight_sd var1_mean var1_sd var2_mean var2_sd var3_mean
<fct> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 ctrl 4.88 0.997 1.5 0.707 22.5 0.707 3.5
2 trt1 4.49 0.453 3.5 0.707 24.5 0.707 5.5
3 trt2 5.72 0.841 5.5 0.707 26.5 0.707 7.5
# ℹ 3 more variables: var3_sd <dbl>, var4_mean <dbl>, var4_sd <dbl>In Ordnung, man hat es geschafft - die dplyr-Einführung ist vorbei. Man kennt jetzt viele der wichtigsten Funktionen des dplyr-Pakets und wie man sie verwendet. Offensichtlich ist es ziemlich überwältigend und niemand verlangt, dass man sich all das auswendig merkt. Stattdessen hoffen wir, dass man sehen kann, wie mächtig diese Funktionen sind und wie sie viel Zeit und manuelle Arbeit sparen können.
Es gibt eine letzte, aber wichtige Information: Sobald man group_by() auf eine Tabelle angewendet hat, bleibt sie gruppiert, es sei denn, man verwendet danach ungroup() darauf. Jede Funktion, die man auf einen Datensatz anwendet, der durch group_by() gegangen ist, wird also separat pro Gruppe angewendet. Das verursachte oben keine Probleme, da wir nie etwas anderes getan haben, als die summarize()-Funktion auf die gruppierten Daten anzuwenden, aber man muss sich dessen bewusst sein, wenn man die gruppierten (Zusammenfassungs-)Ergebnisse für weitere Schritte verwendet. Andernfalls kann das zu unerwarteten Ergebnissen führen. Man kann ein Beispiel und weitere Ressourcen zu solchen unbeabsichtigten Ergebnissen hier finden.
Zusammenfassung
Gut gemacht! Man hat die grundlegenden Tidyverse-Fähigkeiten erworben, auf die Datenwissenschaftler täglich angewiesen sind, um unordentliche Daten in saubere, analysierbare Datensätze zu verwandeln.
Das Tidyverse ist eine Sammlung von R-Paketen, die für die Datenwissenschaft entwickelt wurden und die Datenmanipulation einfacher, schneller und mächtiger macht.
Tibbles sind die moderne Neuvorstellung von data.frames im Tidyverse und bieten verbesserte Anzeigeformatierung und konsistenteres Verhalten.
Der Pipe-Operator (
%>%oder|>) ist ein mächtiges Werkzeug, das Code lesbarer macht, indem er es ermöglicht, Operationen in einer logischen Links-nach-rechts-Sequenz zu verketten.-
Die zentralen dplyr-“Verben” bieten eine konsistente Grammatik für die Datenmanipulation:
-
select(): Bestimmte Spalten nach Name, Position oder Muster auswählen -
filter(): Zeilen extrahieren, die bestimmte Bedingungen erfüllen -
arrange(): Daten basierend auf Spaltenwerten sortieren -
mutate(): Neue Spalten erstellen oder bestehende modifizieren -
summarize(): Zusammenfassende Statistiken berechnen
-
-
Diese Verben werden besonders mächtig, wenn sie kombiniert werden mit:
-
group_by(): Operationen separat innerhalb von Gruppen durchführen -
across(): Dieselbe Funktion auf mehrere Spalten anwenden - Hilfsfunktionen wie
starts_with(),contains()undwhere()
-
Man sollte daran denken,
ungroup()nach gruppierten Operationen zu verwenden, um unerwartete Ergebnisse in nachfolgenden Analyseschritten zu vermeiden.
Fußnoten
Aber es war nicht das erste Paket, das es verwendete. Dieser Blogpost hat eine schöne Zusammenfassung der Geschichte des Pipe-Operators in R.↩︎
Zitat
@online{schmidt2026,
author = {{Dr. Paul Schmidt}},
publisher = {BioMath GmbH},
title = {2. Das Tidyverse},
date = {2026-02-07},
url = {https://biomathcontent.netlify.app/de/content/r_intro/02_tidyverse.html},
langid = {de}
}