Um alle in diesem Kapitel verwendeten Pakete zu installieren und zu laden, führe folgenden Code aus:
Einleitung
Dieses Kapitel führt durch die Erstellung der ggplots aus dem vorherigen Kapitel. Das Ziel ist es, Schritt für Schritt zu erklären, wie diese Plots erstellt wurden, um sowohl die Syntax von ggplot2 als auch die Überlegungen hinter jeder Visualisierungsentscheidung zu verstehen.
Im vorherigen Kapitel haben wir die Beziehung zwischen Düngeranwendung und Ertragssteigerungen analysiert. Nun konzentrieren wir uns darauf, wie man diese Beziehung mit ggplot2, einem der mächtigsten Visualisierungspakete in R, effektiv visualisiert.
Datenvorbereitung
Zuerst importieren wir den gleichen Datensatz, den wir im vorherigen Kapitel verwendet haben, und fitten auch die gleichen Regressionsmodelle. Dadurch haben wir alles vorbereitet, was wir zur Erstellung der Plots benötigen.
Dieser Datensatz enthält Informationen von zwei Landwirten, die unterschiedliche Mengen Dünger angewendet und die resultierenden Ertragssteigerungen aufgezeichnet haben.
Grundlegende ggplot-Struktur
Bevor wir uns unseren spezifischen Plots widmen, verstehen wir die grundlegende Struktur von ggplot2. Alle ggplot2-Visualisierungen folgen einem schichtweisen Ansatz, bei dem man:
- Mit einer grundlegenden
ggplot()-Funktion beginnt, die die Daten und ästhetischen Zuordnungen definiert - Schichten mit dem
+-Operator hinzufügt - Verschiedene Aspekte wie Skalen, Beschriftungen und Themes anpasst
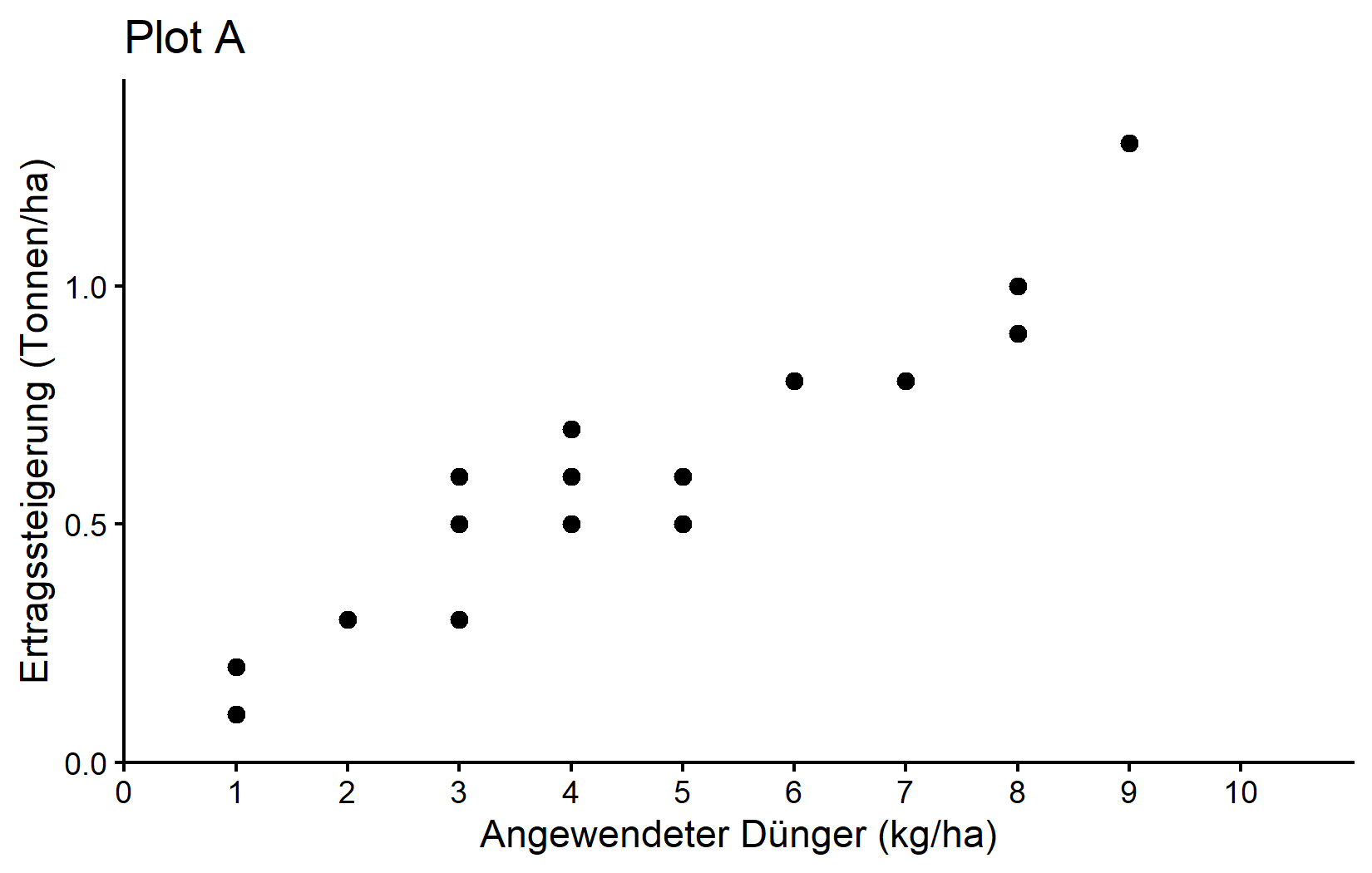
Unser erster Plot: Plot A
Erstellen wir den ersten Plot aus dem Kapitel zu Korrelation & Regression, der die Beziehung zwischen Düngeranwendung und Ertragssteigerung als Streudiagramm zeigt.
Schritt 1: Der minimale Plot
Wir beginnen mit dem minimalen Code, der für die Erstellung eines Streudiagramms erforderlich ist:
ggplot(data = dat) +
aes(x = fert, y = yield_inc) +
geom_point()Schauen wir uns an, was jeder Teil bewirkt:
-
ggplot(data = dat): Dies initialisiert ein ggplot-Objekt und spezifiziert den zu verwendenden Datensatz. -
aes(x = fert, y = yield_inc): Dies definiert die ästhetischen Zuordnungen - welche Variablen auf welche Achsen gehören. -
geom_point(): Dies fügt eine Schicht von Punkten hinzu, um ein Streudiagramm zu erstellen.
Einfach gesagt: Die durch geom_point() erstellten Punkte wissen, wo sie gezeichnet werden sollen, weil die in aes() definierten Ästhetiken ihnen sagen, welche Variablen aus den data für die x- und y-Achsen zu verwenden sind.
Beachte, dass es tatsächlich zwei Möglichkeiten gibt, die ästhetischen Zuordnungen einzuschließen:
# Methode 1: aes() innerhalb von ggplot()
ggplot(data = dat, mapping = aes(x = fert, y = yield_inc)) +
geom_point()
# Methode 2: aes() als separate Schicht
ggplot(data = dat) +
aes(x = fert, y = yield_inc) +
geom_point()Beide Methoden erzeugen identische Plots. In diesem Tutorial verwenden wir die zweite Methode, da sie den Code lesbarer macht, besonders wenn wir mehrere Schichten hinzufügen. Du solltest aber beide Ansätze kennen, um nicht verwirrt zu sein, wenn sie in anderem Code auftreten.
Schritt 2: Anpassung des Punktaussehens
Machen wir die Punkte größer, um die Sichtbarkeit zu verbessern:
ggplot(data = dat) +
aes(x = fert, y = yield_inc) +
geom_point(size = 2)Der Parameter size = 2 vergrößert alle Punkte. Die Standardgröße ist 1,5, also machen wir sie etwas größer.
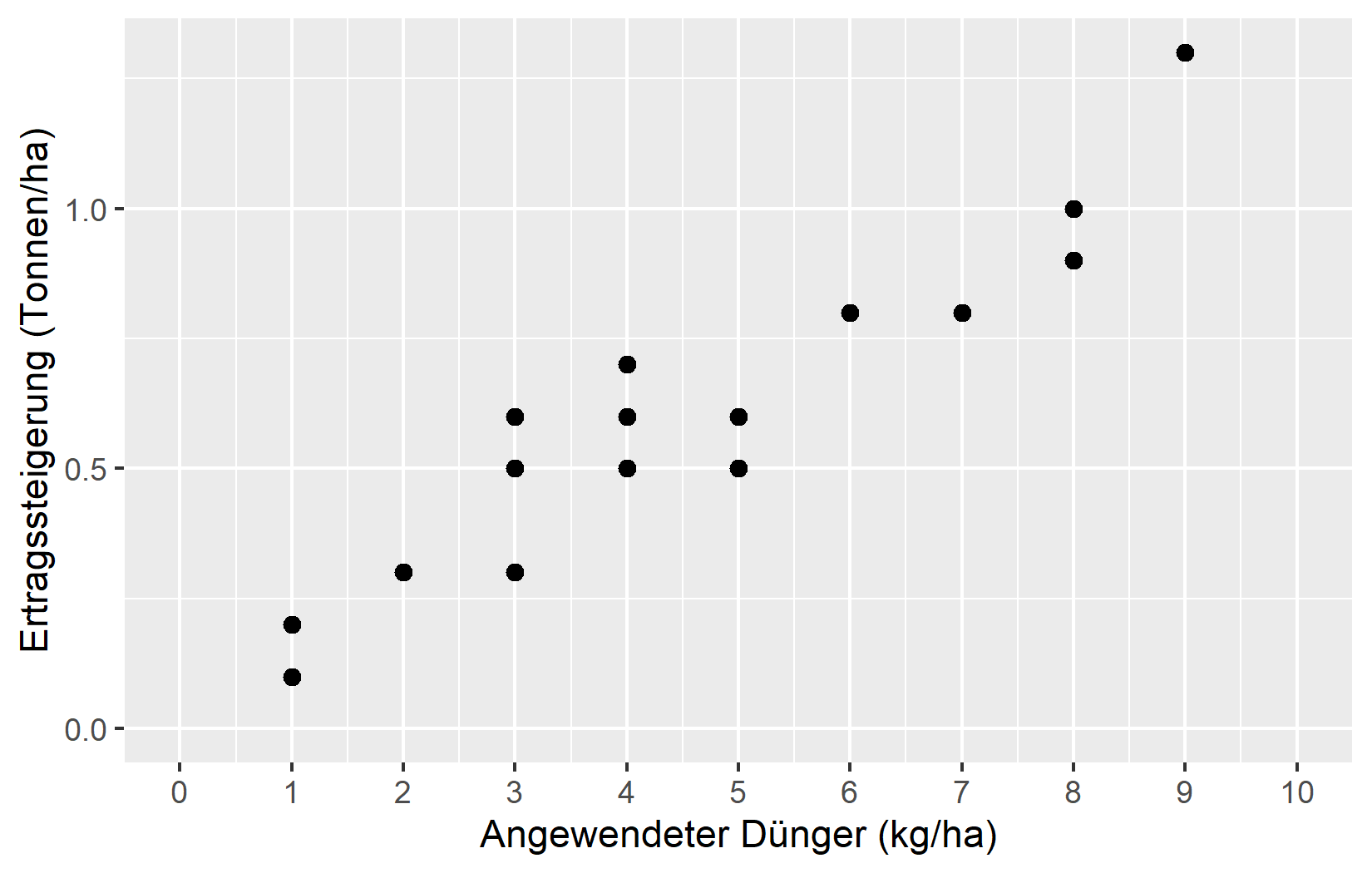
Schritt 3: Verbesserung der Achsenbeschriftungen und -bereiche
Nun passen wir die x- und y-Achsen an, um besseren Kontext zu bieten:
ggplot(data = dat) +
aes(x = fert, y = yield_inc) +
geom_point(size = 2) +
scale_x_continuous(
name = "Angewendeter Dünger (kg/ha)",
limits = c(0, 10),
breaks = seq(0, 10, by = 1)
) +
scale_y_continuous(
name = "Ertragssteigerung (Tonnen/ha)",
limits = c(0, NA)
)Schauen wir uns an, was wir hinzugefügt haben:
-
scale_x_continuous(): Dies passt die x-Achse an, die eine kontinuierliche Variable (Düngeranwendung) darstellt.-
name = "Angewendeter Dünger (kg/ha)": Setzt eine aussagekräftige Achsenbeschriftung mit Einheiten. -
limits = c(0, 10): Setzt den Bereich der x-Achse von 0 bis 10. -
breaks = seq(0, 10, by = 1): Erstellt Teilstriche bei jeder ganzen Zahl von 0 bis 10.
-
-
scale_y_continuous(): Dies passt die y-Achse (Ertragssteigerung) an.-
name = "Ertragssteigerung (Tonnen/ha)": Setzt eine aussagekräftige Achsenbeschriftung mit Einheiten. -
limits = c(0, NA): Setzt die untere Grenze auf 0, lässt aber die obere Grenze beim Standardwert (NA bedeutet “verwende den Standard”).
-
Die y-Achse bei 0 beginnen zu lassen ist gute Praxis für diese Art von Daten, da es die wahre Größenordnung der Ertragssteigerungen ohne Übertreibung zeigt.
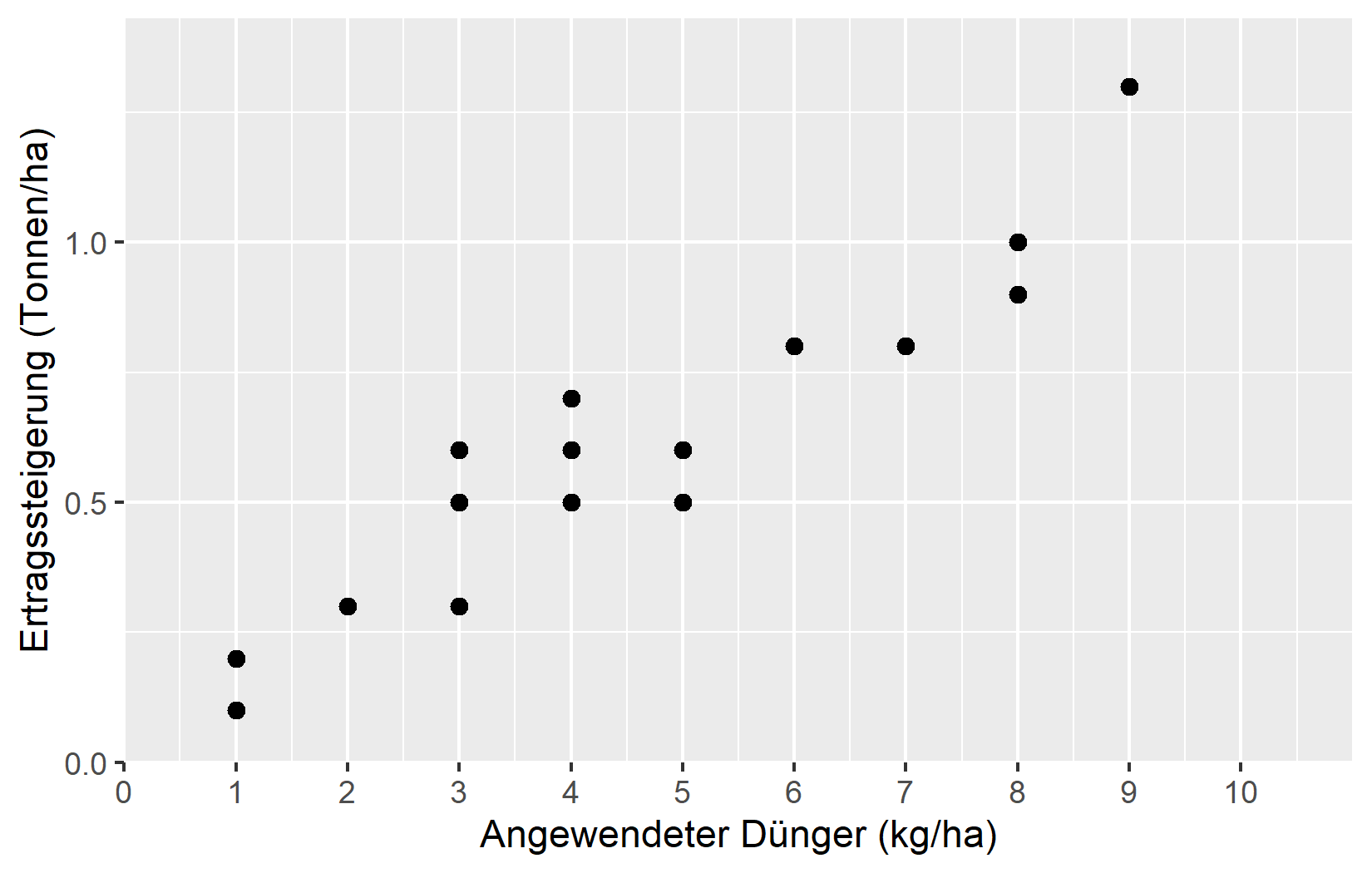
Schritt 4: Verbesserung des visuellen Abstands
Für diesen spezifischen Plot könnte man argumentieren, dass es keinen Sinn macht, Werte unter 0 zu zeigen - zumindest für den angewendeten Dünger, da es nicht möglich ist, eine negative Menge Dünger anzuwenden. Selbst nach dem Setzen der unteren Grenze auf 0 fügt ggplot jedoch etwas zusätzlichen Platz unter dieser Grenze hinzu. Um dieses Standardverhalten zu verhindern, können wir das expand-Argument verwenden. Während wir einfach expand = c(0, 0) setzen könnten, um allen zusätzlichen Platz zu entfernen - sowohl unter der unteren Grenze als auch über der oberen Grenze - ist dies keine elegante Lösung, da dann nicht mehr genug Platz an der oberen Grenze wäre. Stattdessen ist die Verwendung von expand = expansion(mult = c(0, 0.1)) normalerweise eine bessere Lösung, da sie 0% zusätzlichen Platz an der unteren Grenze und 10% zusätzlichen Platz an der oberen Grenze hinzufügt. So können wir sicherstellen, dass der Plot ausgewogen und visuell ansprechend aussieht. Und obwohl expand = expansion(mult = c(0, 0.1)) kryptisch erscheinen mag, ist das Gute daran, dass es in andere Plots mit dem gleichen Problem kopiert werden kann, wenn man keinen zusätzlichen Platz unter 0 möchte.
ggplot(data = dat) +
aes(x = fert, y = yield_inc) +
geom_point(size = 2) +
scale_x_continuous(
name = "Angewendeter Dünger (kg/ha)",
limits = c(0, 10),
breaks = seq(0, 10, by = 1),
expand = expansion(mult = c(0, 0.1))
) +
scale_y_continuous(
name = "Ertragssteigerung (Tonnen/ha)",
limits = c(0, NA),
expand = expansion(mult = c(0, 0.1))
)Schritt 5: Anwenden eines Themes
Schließlich wenden wir ein sauberes Theme auf unseren Plot an:
plotA <- ggplot(data = dat) +
aes(x = fert, y = yield_inc) +
geom_point(size = 2) +
scale_x_continuous(
name = "Angewendeter Dünger (kg/ha)",
limits = c(0, 10),
breaks = seq(0, 10, by = 1),
expand = expansion(mult = c(0, 0.1))
) +
scale_y_continuous(
name = "Ertragssteigerung (Tonnen/ha)",
limits = c(0, NA),
expand = expansion(mult = c(0, 0.1))
) +
theme_classic() +
labs(title = "Plot A")
plotAWir haben hinzugefügt: - theme_classic(): Dies wendet ein sauberes, einfaches Theme mit Achsenlinien aber ohne Gitternetzlinien an. - labs(title = "Plot A"): Dies fügt einen Titel zum Plot hinzu.
Wir haben unseren Plot auch in einer Variable namens plotA gespeichert, damit wir ihn später wiederverwenden können.
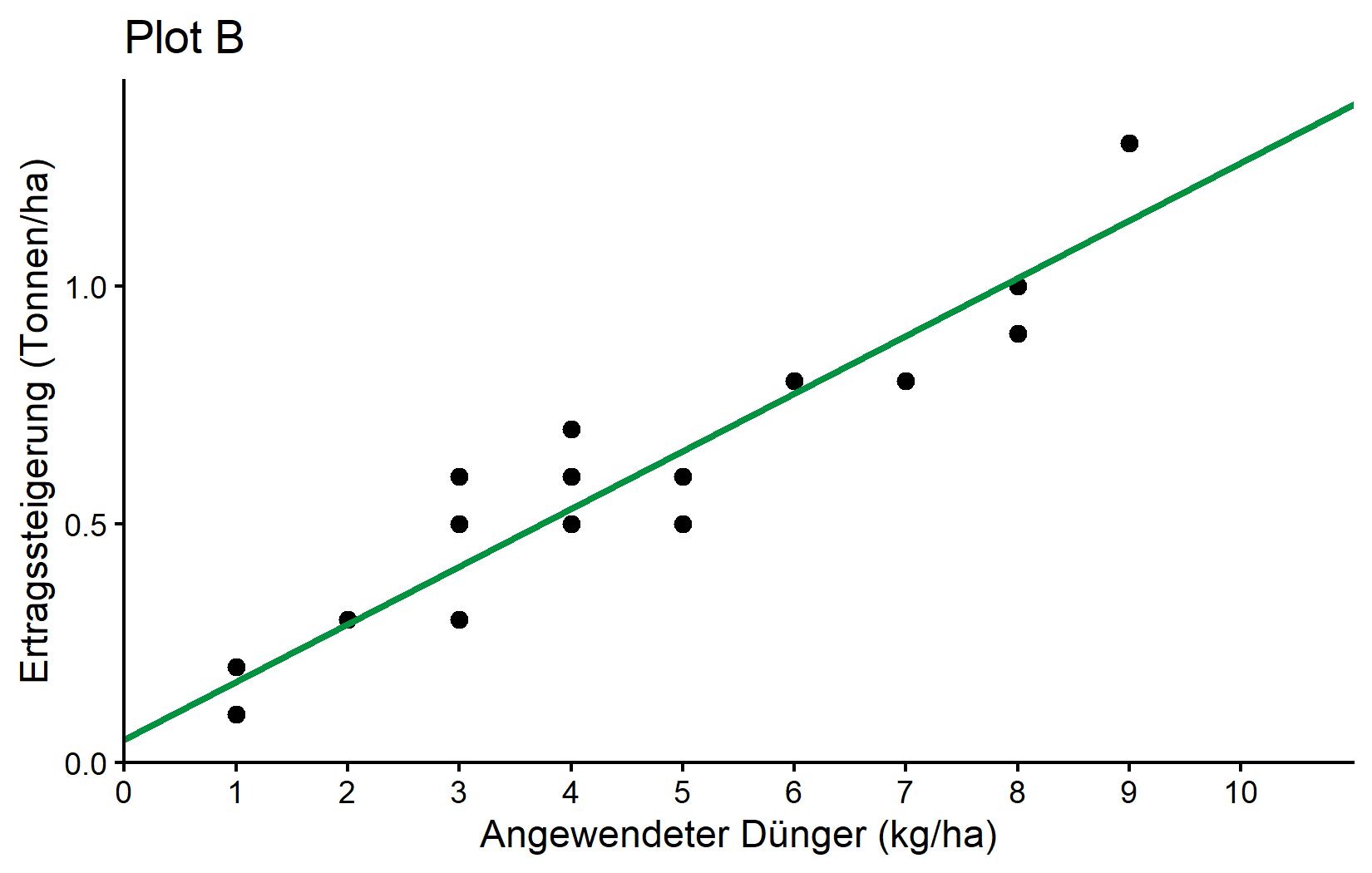
Plot B: Hinzufügen einer Regressionslinie
Für Plot B bauen wir auf Plot A auf, indem wir eine Regressionslinie hinzufügen. Denke daran, dass wir das Ergebnis der linearen Regression in der Variable reg gespeichert haben.
reg
Call:
lm(formula = yield_inc ~ fert, data = dat)
Coefficients:
(Intercept) fert
0.04896 0.12105 Wir könnten nun diese Werte manuell kopieren und in eine zusätzliche geom_abline()-Schicht einfügen:
plotB <- plotA +
geom_abline(
intercept = 0.04896, # Der Achsenabschnitt aus unserer Regression
slope = 0.12105, # Die Steigung aus unserer Regression
color = "#00923f", # Eine grüne Farbe
linewidth = 1 # Etwas dickere Linie
) +
labs(title = "Plot B")
plotBDies ist jedoch keine gute Idee, denn wenn wir später das Regressionsmodell ändern, müssten wir daran denken, auch die Achsenabschnitts- und Steigungswerte im Plot zu aktualisieren. Stattdessen können wir das reg-Objekt direkt verwenden, um die Koeffizienten zu extrahieren:
reg$coefficients(Intercept) fert
0.04896266 0.12104866 Dies gibt uns einen Vektor mit den Achsenabschnitts- und Steigungswerten, die wir in unserer geom_abline()-Schicht verwenden können. So geht’s:
plotB <- plotA +
geom_abline(
intercept = reg$coefficients[1], # Der Achsenabschnitt aus unserer Regression
slope = reg$coefficients[2], # Die Steigung aus unserer Regression
color = "#00923f", # Eine grüne Farbe
linewidth = 1 # Etwas dickere Linie
) +
labs(title = "Plot B")
plotBWas hier neu ist: - geom_abline(): Dies fügt eine gerade Linie mit einem spezifizierten Achsenabschnitt und einer Steigung hinzu. - intercept = reg$coefficients[1]: Verwendet den Achsenabschnitt aus unserem Regressionsmodell. - slope = reg$coefficients[2]: Verwendet die Steigung aus unserem Regressionsmodell. - color = "#00923f": Setzt die Linienfarbe auf einen bestimmten Grünton mit hexadezimalem Farbcode. - linewidth = 1: Setzt die Dicke der Linie (der Standard ist 0,5).
Die Funktion geom_abline() ist perfekt zur Visualisierung unserer Regressionslinie, da sie direkt Achsenabschnitts- und Steigungsparameter akzeptiert. Wir extrahieren diese Werte aus unserem Regressionsmodell mit reg$coefficients.
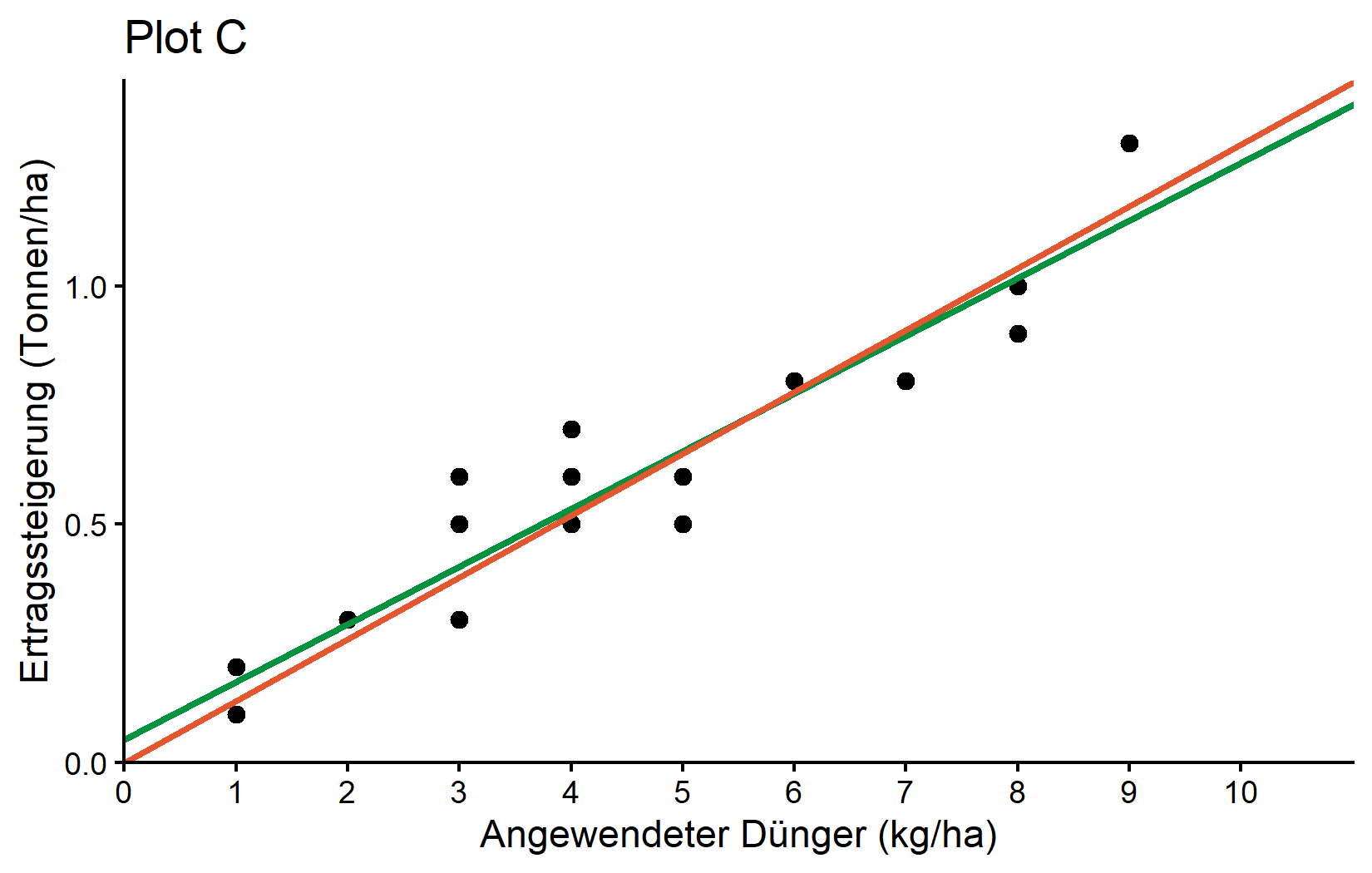
Plot C: Vergleich zweier Regressionslinien
Schließlich erstellen wir Plot C, der beide Regressionsmodelle vergleicht - eines mit Achsenabschnitt und eines ohne. Wir wiederholen im Grunde, was wir gerade gemacht haben, indem wir eine weitere geom_abline()-Schicht hinzufügen, aber diesmal verwenden wir das reg_noint-Objekt, um die Steigung des Modells ohne Achsenabschnitt zu erhalten.
plotC <- plotB +
geom_abline(
intercept = 0,
slope = reg_noint$coefficients[1],
color = "#e4572e",
linewidth = 1
) +
labs(title = "Plot C")
plotCWas in diesem Plot neu ist: - Wir haben eine zweite geom_abline() mit folgenden Eigenschaften hinzugefügt: - intercept = 0: Dies zwingt die Linie, durch den Ursprung zu verlaufen - slope = reg_noint$coefficients[1]: Verwendet die Steigung aus unserem Modell ohne Achsenabschnitt - color = "#e4572e": Verwendet eine orange-rote Farbe, um sie von der ersten Linie zu unterscheiden
Dieser Plot vergleicht effektiv zwei verschiedene Modellierungsansätze - einen, der eine von Null verschiedene Ertragssteigerung erlaubt, wenn kein Dünger angewendet wird (grüne Linie), und einen, der die Linie durch den Ursprung zwingt (orange-rote Linie).
Zusammenfassung
Glückwunsch! Du hast nun gelernt, wie man drei informative Plots erstellt, die die Beziehung zwischen Düngeranwendung und Ertragssteigerung visualisieren. Diese Plots bauten progressiv aufeinander auf, um eine vollständige Geschichte zu erzählen:
Plot A: Zeigte die Rohdaten als Streudiagramm Plot B: Fügte eine Regressionslinie hinzu, um die lineare Beziehung zu visualisieren Plot C: Verglich zwei verschiedene Regressionsansätze
Dabei hast du mehrere wichtige ggplot2-Konzepte gelernt:
- Grundstruktur: Jeder ggplot besteht aus Daten, ästhetischen Zuordnungen und Schichten, die mit dem +-Operator hinzugefügt werden.
- Geometrische Objekte (geoms): Verschiedene geoms wie geom_point() und geom_abline() erstellen verschiedene visuelle Elemente.
- Skalen: Funktionen wie scale_x_continuous() kontrollieren, wie Variablen auf visuelle Eigenschaften abgebildet werden.
- Anpassung: Man kann praktisch jeden Aspekt des Plots kontrollieren, von Achsengrenzen bis zu Farben und Text.
- Themes: Vordefinierte Themes wie theme_classic() setzen schnell den gesamten visuellen Stil.
- Code-Wiederverwendbarkeit: Das Speichern von Plots in Variablen ermöglicht es, sie schrittweise aufzubauen.
Denke daran, dass effektive Datenvisualisierung mehr ist als nur schön aussehende Plots zu erstellen - es geht darum, Erkenntnisse klar zu kommunizieren. Die Entscheidungen, die wir in diesen Plots getroffen haben (Achsen bei Null beginnen lassen, klare Beschriftungen verwenden, informative Regressionslinien hinzufügen), helfen sicherzustellen, dass die Daten genau dargestellt und die Geschichte effektiv erzählt wird.
Für eine umfassendere Einführung in ggplot2 mit detaillierten Beispielen schaue dir an:
- “How I use ggplot2” - Ein Tutorial vom Autor dieses Kurses mit zusätzlichen Techniken und Anpassungsoptionen.
- Die {ggplot2}-Dokumentation für vollständige Referenzinformationen.
Zitat
@online{schmidt2026,
author = {{Dr. Paul Schmidt}},
publisher = {BioMath GmbH},
title = {5. Unsere ersten ggplots},
date = {2026-02-07},
url = {https://biomathcontent.netlify.app/de/content/r_intro/05_firstggplot.html},
langid = {de}
}