Um alle in diesem Kapitel verwendeten Pakete zu installieren und zu laden, führt man folgenden Code aus:
Einführung in R-Projekte
Bevor wir uns mit den Besonderheiten des Importierens und Exportierens von Daten beschäftigen, ist es wichtig zu verstehen, wie R-Projekte das Leben viel einfacher machen können, wenn man mit Dateien außerhalb von R arbeitet.
R-Projekte einrichten
R-Projekte sind eine Funktion von RStudio, die dabei hilft, die Arbeit zu organisieren. Sie halten alle Dateien, die mit einer bestimmten Analyse verbunden sind - Daten, R-Skripte, Ergebnisse, Abbildungen - in einem Verzeichnis zusammen. Das hat mehrere Vorteile:
- Bessere Organisation der Arbeit
- Einfachere Zusammenarbeit mit anderen
- Vereinfachte Pfadbehandlung beim Importieren und Exportieren von Daten
Um ein neues R-Projekt zu erstellen:
- In RStudio geht man zu File -> New Project
- Man wählt entweder “New Directory” oder “Existing Directory” je nach Bedarf. (Wir haben normalerweise bereits vorher einen neuen Ordner erstellt und wählen dann “Existing Directory” in diesem Schritt.)
- Man folgt dem Assistenten, um die Einrichtung abzuschließen
Um zu überprüfen, ob das wie beabsichtigt funktioniert hat, kann man zwei Dinge überprüfen: (1) In der oberen rechten Ecke von RStudio sollte man jetzt den Namen des Ordners sehen, den man als R-Projekt-Ordner ausgewählt hat. (2) Außerhalb von RStudio, im Datei-Explorer, sollte man eine Datei mit der Erweiterung .Rproj in dem ausgewählten Ordner sehen. Diese Datei ist die R-Projekt-Datei und sie enthält alle Informationen über das Projekt.
Die Hauptvorteile, die zusätzlichen Schritte zur Erstellung eines R-Projekts zu unternehmen, sind:
-
Automatisches Arbeitsverzeichnis: Wenn man ein R-Projekt öffnet, wird das sogenannte Arbeitsverzeichnis automatisch auf den Projektordner gesetzt. Das Arbeitsverzeichnis ist im Grunde der Ort/Ordner auf dem Computer, wo R nach zu lesenden Dateien sucht und wo es Dateien speichert. Bevor man ein R-Projekt einrichtet, befindet sich dieses Arbeitsverzeichnis wahrscheinlich an einem zufälligen Ort auf dem Computer - man kann das herausfinden, indem man den Code
getwd()in der Konsole ausführt. - Projektverwaltung: R-Projekte helfen dabei, Dateien und Skripte auf strukturierte Weise zu verwalten. Man kann einfach zwischen Projekten wechseln, ohne sich um Dateipfade oder Arbeitsverzeichnisse sorgen zu müssen. Ein Projekt merkt sich sogar, welche R-Skripte geöffnet waren, als man das Projekt zuletzt geschlossen hat, sodass man genau dort weitermachen kann, wo man aufgehört hat. Sogar das Teilen eines ganzen Projekts mit jemand anderem ist einfach, da man einfach den gesamten Ordner senden kann und sie ihn in RStudio öffnen können. Das ist viel einfacher, als ihnen eine Reihe von Dateien zu senden und ihnen zu sagen, sie sollen sie an die richtige Stelle auf ihrem Computer legen.
- Versionskontrolle: Etwas Fortgeschritteneres wäre die Integration mit Versionskontroll-Systemen wie Git, was es einfacher macht, Änderungen zu verfolgen und mit anderen zusammenzuarbeiten. Wir werden das jedoch in diesem Einführungskurs nicht behandeln.
Schau dir die offizielle RStudio-Erklärung zu Using RStudio Projects und Kapitel 1.6 Projects in RStudio im Buch “An Introduction to R” an
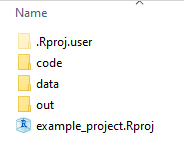
Organisation mit Unterordnern
Es ist nicht unbedingt notwendig, aber eine gute Praxis, den R-Projekt-Ordner mit einer konsistenten Ordnerstruktur zu organisieren, d.h. mit Unterordnern wie
-
data/: Für Roh- und verarbeitete Datendateien -
code/oderR/: Für R-Skripte und Funktionen -
out/oderresults/: Für Ausgaben wie Diagramme, Tabellen und Analyseergebnisse
Diese Organisation macht es einfacher, Dateien zu finden und einen sauberen Arbeitsablauf zu erhalten.
Von diesem Moment an wird der Code, den man sieht, nur funktionieren, wenn man ein R-Projekt erstellt hat und die Unterordner data/, out/ und code/ darin erstellt hat. Der Name des Hauptordners (d.h. R-Projekt-Name) kann also alles sein, was man gewählt hat, aber die Namen der Unterordner und Dateien innerhalb der Unterordner müssen auf dem PC identisch mit denen hier sein, damit der Code funktioniert. Wenn man andere Namen verwenden möchte, kann man das tun, aber dann muss man den Code entsprechend ändern. Hier ist ein Screenshot davon, wie es in deinem R-Projekt-Ordner gerade aussehen sollte:
Außerdem (!) muss man die Dateien herunterladen, die im data-Ordner hier auf GitHub verfügbar sind (Download-Link). Diese Dateien sollten dann in deinem data-Ordner gespeichert werden:
- an_excel_file.xlsx
- Clewer&Scarisbrick2001.csv
- Mead1993.csv
- vision fixed.xls
- vision.xls
- yield_increase.csv
Das {here}-Paket
Wieder - nicht unbedingt notwendig, aber eine gute Praxis: Das {here}-Paket ist ein großartiges Werkzeug zur Verwaltung von Dateipfaden in R-Projekten. Es erkennt automatisch das Projektverzeichnis und ermöglicht es, Dateipfade relativ dazu zu erstellen, was den Code portabler und einfacher mit anderen zu teilen macht. Angenommen, es gäbe zum Beispiel eine Datei mydata.xlsx im data-Unterordner, könnte man einfach schreiben
here("data", "mydata.xlsx")Außerdem ist das {here}-Paket sehr nützlich zum Erstellen von Dateipfaden, die plattformunabhängig sind. Das bedeutet, dass man denselben Code auf verschiedenen Betriebssystemen (Windows, Mac, Linux) verwenden kann, ohne sich um Unterschiede in der Dateipfad-Syntax sorgen zu müssen.
CSV
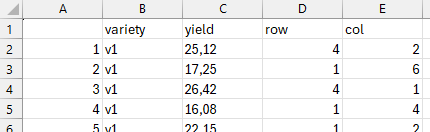
Eine der einfachsten Möglichkeiten, mit dem Datenimport zu beginnen, ist das direkte Lesen von einer URL. Dieser Ansatz erfordert nicht, dass man Dateien herunterlädt und würde auch ohne ein eingerichtetes R-Projekt funktionieren. Man kann zum Beispiel die folgende CSV-Datei so importieren:
variety yield row col
1 v1 25.12 4 2
2 v1 17.25 1 6
3 v1 26.42 4 1Wenn also die Internetverbindung funktioniert, kann man diesen Code überall ausführen und er wird funktionieren. Zuerst speichern wir die URL, wo die Datei gespeichert ist (als String mit ") in einer Variable x. Dann übergeben wir diese Variable an die Importfunktion read.csv(). Man könnte natürlich auch einfach die URL direkt in die Funktion einfügen.
Man beachte, dass man diese URL auch einfach in den Browser einfügen und die Datei sehen könnte - es ist wirklich nur eine CSV-Datei, aber anstatt auf dem Computer zu sein, ist sie im Internet.
Importieren
Außerdem sehen wir, dass die Funktion, die das Importieren übernommen hat, read.csv() heißt. Das ist eine Funktion, die Teil von base R ist, was bedeutet, dass sie eingebaut ist und keine zusätzlichen Pakete erfordert. Die meisten Importfunktionen in R beginnen mit read. und enden mit dem Dateityp, den sie zum Lesen/Importieren entwickelt wurden. CSV ist die Abkürzung für “Comma-Separated Values” und ist ein gängiges Format zur Speicherung tabellarischer Daten. Es ist eine reine Textdatei, die (normalerweise) Kommas zur Trennung von Werten verwendet - man kann gerne der obigen URL folgen und sich die Daten tatsächlich ansehen. Man wird feststellen, dass sie tatsächlich einfach alle Datenpunkte pro Zeile enthält, getrennt durch Kommas.
Nach unserem erfolgreichen Import können wir sehen, dass die Daten jetzt in einer Variable namens mydata gespeichert sind. Diese Variable ist ein data frame, weil read.csv() eine baseR-Funktion ist und daher in den baseR-Datentyp für Tabellen importiert: data.frame. Man beachte, dass das Tidyverse auch sein eigenes Paket nur zum Importieren von Daten hat, das readr genannt wird. Dieses Paket ist Teil des Tidyverse und ist darauf ausgelegt, schneller und benutzerfreundlicher als base R-Funktionen zu sein. Um stattdessen seine Funktion zu verwenden, müsste man die Funktion read_csv() anstelle von read.csv() verwenden. Wie man erwartet haben könnte, importiert sie in den Tidyverse-Datentyp für Tabellen: tibble. Außerdem zeigt sie auch einige zusätzliche Informationen über die Daten beim Importieren:
mytbl <- read_csv(file = x) # Daten importierenRows: 24 Columns: 4
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): variety
dbl (3): yield, row, col
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.head(mytbl, n = 3) # erste Zeilen der Daten anzeigen# A tibble: 3 × 4
variety yield row col
<chr> <dbl> <dbl> <dbl>
1 v1 25.1 4 2
2 v1 17.2 1 6
3 v1 26.4 4 1Man beachte, dass in unserem Fall die einzige Information, die die Funktionen zum Importieren dieses Datensatzes benötigen, sein Ort/Pfad ist. Dieser Pfad kann entweder eine URL wie oben oder ein Dateipfad auf dem Computer sein. Es gibt jedoch mehrere Argumente, die man zur Funktion hinzufügen kann, um den Importprozess anzupassen. Man kann zum Beispiel angeben, ob die erste Zeile der Datei Spaltennamen enthält oder nicht, welches Zeichen/Symbol zur Trennung von Werten verwendet wird, wie mit fehlenden Werten umgegangen werden soll oder sogar ob die ersten paar Zeilen in den Daten übersprungen werden sollen. All dies kann einem ersparen, die Daten vor dem Importieren manuell korrigieren zu müssen. Man kann mehr herausfinden, indem man ?read.csv() oder ?read_csv() ausführt und die Hilfeseite durchgeht.
Exportieren
Wie man vielleicht erraten hat, heißt die Funktion zum Exportieren von Daten write.csv(). Im Gegensatz zu den Importfunktionen benötigt sie mindestens zwei Informationen: Die Daten, die man exportieren möchte, und den Ort/Pfad, wo man sie speichern möchte. Die Funktion erstellt dann eine CSV-Datei an diesem Ort. Um zum Beispiel unsere Tabelle mytbl zu exportieren, würden wir folgenden Code ausführen:
Wie man sehen kann, verwenden wir die here-Funktion, die wir früher eingeführt haben, um den Dateipfad zu erstellen. Das Ausführen davon erstellt eine CSV-Datei namens mytbl.csv im data-Unterordner des R-Projekt-Ordners. Wieder gibt es mehrere zusätzliche Argumente, die man zur Funktion hinzufügen kann, um den Exportprozess anzupassen. Man kann zum Beispiel angeben, ob Zeilennamen eingeschlossen werden sollen oder nicht, indem man das Argument row.names = FALSE setzt. Standardmäßig ist dies auf TRUE gesetzt, sodass Zeilennamen exportiert werden, wie man in den Daten (oder den Screenshots der Daten unten) sehen kann.
Herzlichen Glückwunsch, man hat jetzt erfolgreich Daten in R importiert und exportiert! Das meiste, was jetzt folgt, sind nur verschiedene Versionen desselben Prozesses.
Hier ist eine ziemlich irritierende Tatsache über CSV-Dateien und Microsoft Excel. Obwohl wir gerade eine perfekt funktionierende CSV-Datei exportiert haben, bemerkt man, dass wenn man sie in Excel öffnet, sie möglicherweise nicht gut aussieht, da die Spalten nicht korrekt getrennt sind:
Das liegt daran, dass Excel in vielen regionalen Einstellungen Semikolons (;) anstelle von Kommas (,) als Trennzeichen verwendet, obwohl CSV für “Comma-Separated Values” steht. Das macht zum Beispiel für Deutschland Sinn, wo ein Komma als Dezimaltrennzeichen verwendet wird: Ein halber ist 0,5 anstatt 0.5, also wenn man ein Komma als Trennzeichen verwenden würde, würde es nicht richtig funktionieren.
Wenn man also eine CSV-Datei erstellt, die in Excel (in der jeweiligen regionalen Einstellung) geöffnet werden muss, kann man die Funktion write.csv2() anstelle von write.csv() verwenden. Diese Funktion verwendet Semikolons als Trennzeichen und erstellt eine CSV-Datei, die sich korrekt in Excel öffnet. Man kann auch die read.csv2()/read_csv2()-Funktionen verwenden, um solche Dateien zu importieren.
write.csv2(
x = mytbl,
file = here("data", "mytbl.csv")
)TXT
Textdateien (.txt) sind CSV-Dateien sehr ähnlich, mit dem Hauptunterschied, dass sie oft ein Tabulatorzeichen (Tabulator) als Trennzeichen anstelle eines Kommas verwenden. Im Wesentlichen sind beide reine Textformate, die tabellarische Daten speichern - sie verwenden nur unterschiedliche Zeichen zur Trennung der Werte.
Um eine tabulator-getrennte Textdatei zu importieren, können wir die allgemeinere read.delim()-Funktion in base R oder read_delim() im Tidyverse verwenden. Man beachte, dass die folgenden Codes nur Beispiele sind und nicht funktionieren werden, es sei denn, man hat eine Datei namens mydata.txt im data-Unterordner des R-Projekt-Ordners.
# Base R-Ansatz
txt_data_base <- read.delim(file = here("data", "mydata.txt"), sep = "\t")
# Tidyverse-Ansatz
txt_data_tidy <- read_delim(file = here("data", "mydata.txt"), delim = "\t")Das Exportieren von Textdateien folgt einem ähnlichen Muster wie CSV-Dateien:
# Base R-Ansatz
write.table(x = mytbl,
file = here("data", "mytbl.txt"),
sep = "\t")
# Tidyverse-Ansatz
write_delim(x = mytbl,
file = here("data", "mytbl.txt"),
delim = "\t")Die delim- und sep-Argumente ermöglichen es zu spezifizieren, welches Zeichen Werte in der Ausgabedatei trennen soll. Häufige Trennzeichen sind Tabulatoren ("\t"), Kommas (","), Semikolons (";") und sogar Leerzeichen (" ").
Da CSV- und TXT-Dateien beide im Wesentlichen reine Textformate mit unterschiedlichen Trennzeichen sind, gelten für beide dieselben Prinzipien. Nachdem wir diese einfachen textbasierten Formate behandelt haben, gehen wir nun zur Arbeit mit komplexeren Dateitypen wie Excel über.
Excel
Excel-Dateien mit {readxl} importieren
Excel-Dateien (.xlsx, .xls) werden in vielen Bereichen sehr häufig verwendet. Sie unterstützen mehrere Arbeitsblätter, Formatierung, Formeln und vieles mehr. Um Excel-Dateien in R zu importieren, verwenden wir das {readxl}-Paket, das wir bereits am Anfang dieses Kapitels geladen haben.
Bitte stelle sicher, dass du die Excel-Datei an_excel_file.xlsx von der Kurs-Website heruntergeladen und im data-Unterordner deines R-Projekt-Ordners platziert haben. Andernfalls wird der Code unten nicht funktionieren.
xlsx_path <- here("data", "an_excel_file.xlsx")dat_sheet1 <- read_excel(path = xlsx_path)
dat_sheet1# A tibble: 3 × 2
Name Value
<chr> <dbl>
1 A 12
2 B 13
3 C 12Diese Funktion versucht standardmäßig, das erste Blatt in der Excel-Datei zu lesen. Wenn die Daten in einem anderen Blatt sind, muss man es spezifizieren. Wir können alle Blattnamen in der Datei mit der excel_sheets()-Funktion herausfinden:
excel_sheets(path = xlsx_path)[1] "data1" "otherdata"und dann das zweite Blatt entweder nach Namen oder nach Index importieren:
# Ein spezifisches Blatt nach Namen lesen
dat_sheet2 <- read_excel(path = xlsx_path, sheet = "otherdata")
# Oder nach Blatt-Index
dat_sheet2 <- read_excel(path = xlsx_path, sheet = 2)
dat_sheet2Ähnlich wie bei CSV-Importen kann man anpassen, wie die Daten mit zusätzlichen Argumenten wie col_names, na usw. importiert werden. Man kann sogar einen spezifischen Bereich von Zellen zum Importieren über z.B. range = "A1:C10" auswählen. Schau dir die Dokumentation mit ?read_excel für weitere Details an.
Exportieren nach Excel mit {openxlsx}
Zum Exportieren von Daten in das Excel-Format verwenden wir das {openxlsx}-Paket, das wir am Anfang geladen haben. Dieses Paket bietet zwei Hauptansätze: eine einfache Ein-Zeilen-Funktion und einen detaillierteren Ansatz für größere Kontrolle. Und ja, wir verwenden hier zwei verschiedene Pakete: readxl zum Importieren und openxlsx zum Exportieren von Excel-Dateien.
Eine Tabelle zu einem Blatt
Der einfachste Weg, einen data.frame nach Excel zu exportieren, ist:
write.xlsx(x = mytbl, file = here("data", "exported_data.xlsx"))Wie man sehen kann, fühlt sich das sehr ähnlich an wie write.csv(), write_delim() usw., was schön ist.
Mehrere Tabellen zu mehreren Blättern
Für mehr Kontrolle über das Aussehen der Excel-Datei kann man einen detaillierteren Ansatz verwenden. Nehmen wir an, wir möchten eine Excel-Datei mit zwei Blättern namens “SheetA” und “SheetB” erstellen. Das erste soll mytbl enthalten, während das zweite die PlantGrowth-Daten enthalten soll. Der minimale Code dafür würde so aussehen:
# Eine neue Arbeitsmappe erstellen
mywb <- createWorkbook()
# Blatt A
addWorksheet(wb = mywb, sheetName = "SheetA")
writeData(wb = mywb, sheet = "SheetA", x = mytbl)
# Blatt B
addWorksheet(wb = mywb, sheetName = "SheetB")
writeData(wb = mywb, sheet = "SheetB", x = PlantGrowth)
# Die Arbeitsmappe speichern
saveWorkbook(wb = mywb, here("data", "formatted_excel.xlsx"), overwrite = TRUE)Wie man sehen kann, erstellen wir zuerst eine sogenannte Arbeitsmappe, fügen dann Blätter hinzu und schreiben Daten in diese Blätter. Schließlich speichern wir die Arbeitsmappe in eine Datei. Das Argument overwrite = TRUE ermöglicht es, eine bestehende Datei mit demselben Namen zu überschreiben.
Man beachte, dass das bei weitem nicht alles ist, was man mit dem openxlsx-Paket erreichen kann. Man kann auch Zellen formatieren, Diagramme hinzufügen und vieles mehr. Schauen dir die Dokumentation für weitere Details an.
Andere Dateiformate
Während CSV und Excel die häufigsten Dateiformate sind, kann man in der Arbeit auf andere Typen stoßen. Hier sind einige Pakete zur Behandlung dieser:
-
Statistische Software: Das
haven-Paket kann SPSS (.sav), SAS (.sas7bdat) und Stata (.dta) Dateien importieren/exportieren.
Datenbanken: Pakete wie
DBI,RSQLiteundRMySQLbieten Verbindungen zu verschiedenen Datenbanksystemen.JSON & XML: Die Pakete
jsonliteundxml2behandeln diese web-orientierten Formate.Spezialisierte Formate: Für bereichsspezifische Formate sucht man auf CRAN nach geeigneten Paketen - es gibt wahrscheinlich eine Lösung für die jeweiligen Bedürfnisse.
In den meisten Fällen folgen die Funktionen zum Importieren/Exportieren dieser Formate ähnlichen Mustern wie das, was wir mit CSV und Excel gesehen haben, beginnend mit read_ oder write_ gefolgt vom Formatnamen.
Diagramme exportieren mit ggsave()
Wir wissen, dass wir eigentlich noch nicht gelernt haben, wie man ein ggplot erstellt, aber da wir über Import und Export sprechen, werden wir auch behandeln, wie man Diagramme exportiert.
Während RStudios “Export”-Button im Plots-Panel funktioniert, bietet die ggsave()-Funktion einen reproduzierbareren Ansatz.
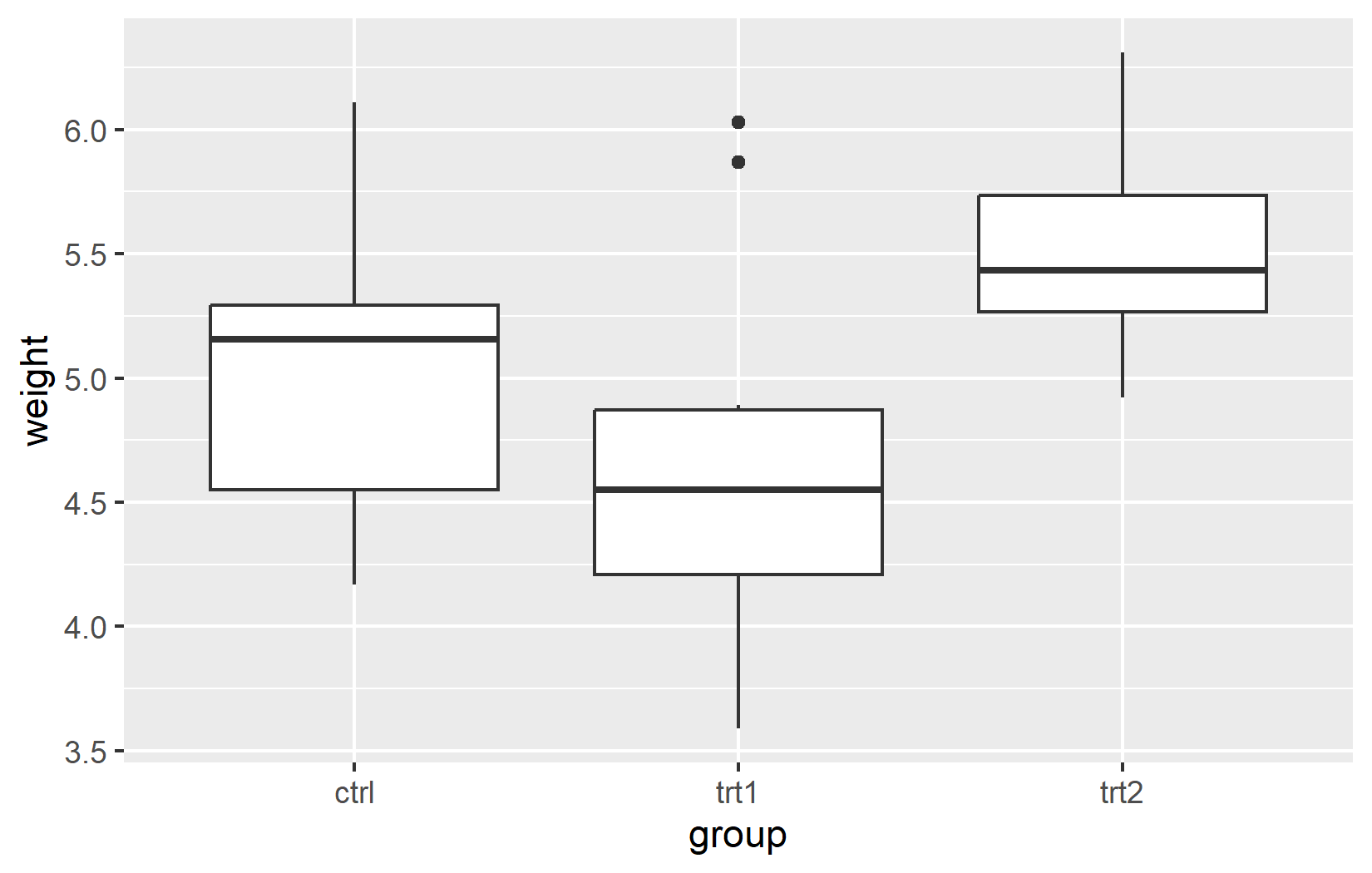
Erstellen wir zuerst ein einfaches Diagramm zur Demonstration, wieder mit dem PlantGrowth-Datensatz:
# Ein Beispieldiagramm erstellen
myplot <- ggplot(data = PlantGrowth, aes(x = group, y = weight)) +
geom_boxplot()
# Das Diagramm anzeigen
myplotJetzt können wir dieses Diagramm mit ggsave() exportieren:
Die ggsave()-Funktion hat mehrere wichtige Argumente:
- filename: Der Name der Datei (einschließlich Erweiterung)
- plot: Das zu speichernde Diagrammobjekt (standardmäßig das zuletzt angezeigte Diagramm, wenn nicht spezifiziert)
- path: Wo die Datei gespeichert werden soll
- width, height: Dimensionen des Bildes
- units: Einheit für Breite/Höhe (“cm”, “in”, “mm”, usw.)
- dpi: Auflösung in Punkten pro Zoll (höher = bessere Qualität aber größere Datei)
Die Dateierweiterung im Dateinamen bestimmt das Ausgabeformat. Oben haben wir ein PNG exportiert, aber wir können genauso gut ein PDF oder SVG exportieren, die Vektorformate sind. Vektorformate sind ideal für publikationsqualitative Abbildungen, da sie ohne Qualitätsverlust vergrößert werden können. Das einzige, was man ändern muss, ist die Dateierweiterung im filename-Argument.
Bei der Wahl eines Dateiformats für Diagramme:
- PNG (.png): Gut für Präsentationen und Web-Verwendung
- JPEG (.jpg): Kleinere Dateigröße aber geringere Qualität
- PDF (.pdf): Vektorformat ideal für Publikationen und Druck
- SVG (.svg): Vektorformat gut für Web-Verwendung und weitere Bearbeitung
- TIFF (.tiff): Hochqualitätsformat oft von Zeitschriften gefordert
Für publikationsqualitative Abbildungen werden normalerweise PDF oder TIFF bevorzugt. Für Präsentationen oder Web-Verwendung funktioniert PNG oft am besten.
Schnellere Alternativen für große Daten
Während die Funktionen, die wir bisher behandelt haben, für die meisten alltäglichen Datenanalysen vollkommen ausreichend sind, kann es bei sehr großen Datensätzen (mit Millionen von Zeilen) vorkommen, dass der Import merklich lange dauert. Für solche Fälle gibt es spezialisierte Pakete, die darauf ausgelegt sind, Daten deutlich schneller zu lesen.
Das {data.table}-Paket
Das {data.table}-Paket ist bekannt für seine extrem schnelle fread()-Funktion (kurz für “fast read”). Diese Funktion kann CSV-Dateien oft 5-10 mal schneller importieren als die Standard-Funktionen:
# data.table installieren und laden falls nötig
# install.packages("data.table")
library(data.table)
# Sehr schneller Import von CSV-Dateien
fast_data <- fread(file = "some_large_dataset.csv")Zusätzlich zum schnellen Import erstellt fread() ein data.table-Objekt, das auch für sehr schnelle Datenmanipulation optimiert ist. Wenn man die Daten jedoch als normalen data.frame oder tibble benötigt, kann man sie einfach konvertieren.
Das {vroom}-Paket
Das {vroom}-Paket ist eine weitere Alternative, die Teil des erweiterten Tidyverse-Ökosystems ist. Es ist besonders darauf spezialisiert, sehr große Dateien extrem schnell zu lesen:
Das Besondere an vroom() ist, dass es einen sogenannten “lazy loading”-Ansatz verwendet - es liest nicht sofort alle Daten in den Speicher, sondern nur die Teile, die man tatsächlich verwendet. Das kann bei gigantischen Datensätzen von Vorteil sein.
Für normale Datenanalyseprojekte mit Datensätzen, die weniger als 100.000 Zeilen haben, sind die Standard-Importfunktionen völlig ausreichend. Die spezialisierten Geschwindigkeitspakete werden erst bei sehr großen Daten (Millionen von Zeilen) oder bei wiederholten Importen derselben großen Datei wirklich nützlich.
Wenn man sich für diese leistungsstarken Alternativen interessiert, sollte man sich die jeweilige Paket-Dokumentation ansehen, da sie auch erweiterte Funktionen für die Datenmanipulation bieten, die über den reinen Import hinausgehen.
Zusammenfassung
Man weiß jetzt, wie man Daten effizient in R hinein- und herausbewegt, eine grundlegende Fähigkeit, die einem unzählige Stunden in der Datenanalyse-Reise sparen wird.
R-Projekte bieten ein robustes Framework zur Organisation der Arbeit, Verwaltung von Dateipfaden und Gewährleistung der Reproduzierbarkeit.
-
Für Datenimport/-export:
- CSV-Dateien:
read.csv()/write.csv()(base R) oderread_csv()/write_csv()(tidyverse) verwenden - TXT-Dateien:
read.delim()/write.table()(base R) oderread_delim()/write_delim()(tidyverse) verwenden - Excel-Dateien:
read_excel()aus {readxl} für Import undwrite.xlsx()aus {openxlsx} für Export verwenden
- CSV-Dateien:
Das {here}-Paket vereinfacht die Dateipfad-Verwaltung und macht den Code portabler und einfacher zu teilen.
-
Bei der Arbeit mit Excel-Dateien sollte man beachten, dass man:
- Spezifische Blätter nach Namen oder Index lesen kann
- Mehrere Tabellen in verschiedene Blätter innerhalb derselben Arbeitsmappe exportieren kann
Für Diagramme verwendet man
ggsave(), um in verschiedenen Formaten (PNG, PDF, SVG) mit präziser Kontrolle über Dimensionen und Qualität zu exportieren.Man sollte das Projekt immer mit einer konsistenten Ordnerstruktur (data/, code/, out/) organisieren, um einen sauberen Arbeitsablauf zu erhalten.
Zitat
@online{schmidt2026,
author = {{Dr. Paul Schmidt}},
publisher = {BioMath GmbH},
title = {3. Datenimport und -export},
date = {2026-02-07},
url = {https://biomathcontent.netlify.app/de/content/r_intro/03_importexport.html},
langid = {de}
}