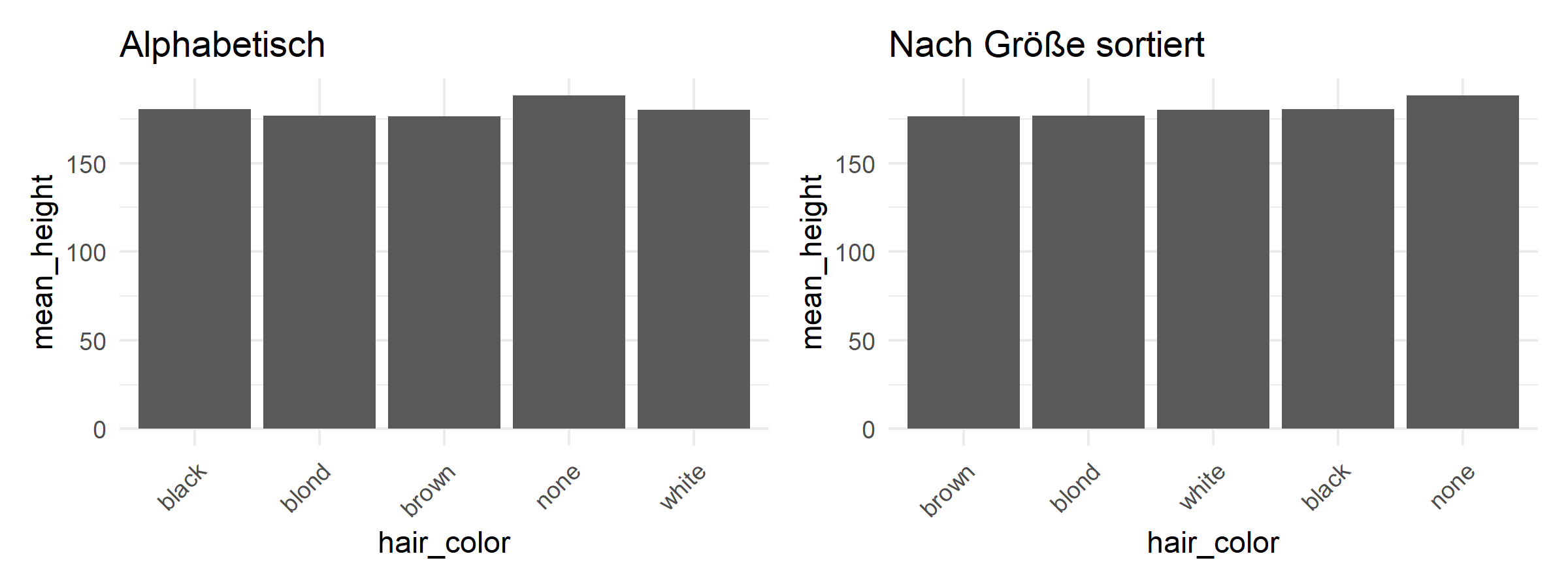Um alle in diesem Kapitel verwendeten Pakete zu installieren und zu laden, führt man folgenden Code aus:
Einleitung
Wer in R mit kategorialen Daten arbeitet, stößt früher oder später auf das Konzept der Faktoren. Für Einsteiger sind sie oft verwirrend: Warum verhält sich eine Spalte plötzlich anders als erwartet? Warum erscheinen die Balken im Diagramm in einer seltsamen Reihenfolge?
Dieses Kapitel erklärt, was Faktoren sind, wann man sie braucht und wie man sie mit dem {forcats}-Paket elegant manipuliert.
Beispieldaten
Wir verwenden wieder den starwars-Datensatz, gefiltert auf Menschen:
# A tibble: 35 × 6
name height mass hair_color eye_color gender
<chr> <int> <dbl> <chr> <chr> <chr>
1 Luke Skywalker 172 77 blond blue masculine
2 Darth Vader 202 136 none yellow masculine
3 Leia Organa 150 49 brown brown feminine
4 Owen Lars 178 120 brown, grey blue masculine
5 Beru Whitesun Lars 165 75 brown blue feminine
6 Biggs Darklighter 183 84 black brown masculine
7 Obi-Wan Kenobi 182 77 auburn, white blue-gray masculine
8 Anakin Skywalker 188 84 blond blue masculine
9 Wilhuff Tarkin 180 NA auburn, grey blue masculine
10 Han Solo 180 80 brown brown masculine
# ℹ 25 more rowsCharacter vs. Factor: Der Unterschied
Character: Einfach Text
Eine Character-Variable ist schlicht Text. R behandelt jeden Wert als eigenständigen String:
# eye_color ist ein Character-Vektor
class(humans$eye_color)[1] "character"# Unique Werte (in der Reihenfolge des ersten Auftretens)
unique(humans$eye_color)[1] "blue" "yellow" "brown" "blue-gray" "hazel" "dark"
[7] "unknown" Wenn wir Character-Werte sortieren, geschieht das alphabetisch:
Factor: Text mit Struktur
Ein Factor ist Text plus zusätzliche Information:
- Levels: Die möglichen Kategorien
- Reihenfolge: Die Sortierung der Levels
[1] "factor"levels(eye_factor) # Die gespeicherten Levels[1] "blue" "blue-gray" "brown" "dark" "hazel" "unknown"
[7] "yellow" Die Levels sind standardmäßig alphabetisch sortiert. Aber wir können eine eigene Reihenfolge definieren:
Warum ist das wichtig?
Die Level-Reihenfolge beeinflusst:
- Die Sortierung in Tabellen
- Die Reihenfolge in Grafiken (z.B. Balkendiagramme)
- Die Referenzkategorie in statistischen Modellen
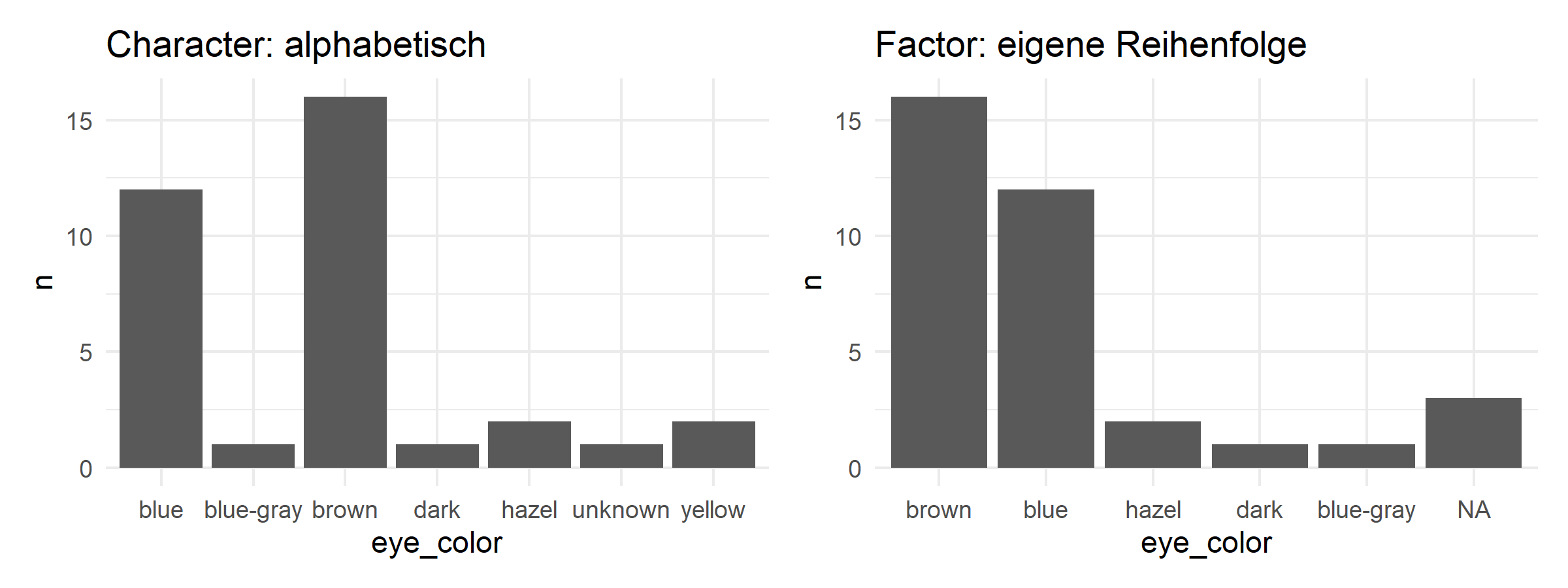
# Mit Character: alphabetische Reihenfolge
p1 <- humans %>%
count(eye_color) %>%
ggplot(aes(x = eye_color, y = n)) +
geom_col() +
labs(title = "Character: alphabetisch") +
theme_minimal()
# Mit Factor: unsere Reihenfolge
p2 <- humans %>%
mutate(eye_color = factor(eye_color,
levels = c("brown", "blue", "hazel", "dark", "blue-gray"))) %>%
count(eye_color) %>%
ggplot(aes(x = eye_color, y = n)) +
geom_col() +
labs(title = "Factor: eigene Reihenfolge") +
theme_minimal()
# Nebeneinander anzeigen
p1 + p2a) Prüfe mit class(), ob hair_color im humans-Datensatz ein Character oder Factor ist.
b) Wandle hair_color in einen Factor um und zeige die Levels an.
c) Erstelle einen Factor für hair_color mit der Reihenfolge: “brown”, “black”, “blond”, “auburn”, dann alle anderen.
# a) Klasse prüfen
class(humans$hair_color)[1] "character" [1] "auburn" "auburn, grey" "auburn, white" "black"
[5] "blond" "brown" "brown, grey" "grey"
[9] "none" "white" [1] "brown" "black" "blond" "auburn"
[5] "auburn, grey" "auburn, white" "grey" "white"
[9] "none" Faktoren erstellen
factor() vs. as_factor()
Es gibt zwei Hauptfunktionen zum Erstellen von Faktoren:
[1] rot blau rot grün blau
Levels: blau grün rot# as_factor(): Levels nach Reihenfolge des ersten Auftretens
as_factor(farben)[1] rot blau rot grün blau
Levels: rot blau grün| Funktion | Paket | Level-Reihenfolge |
|---|---|---|
factor() |
base R | Alphabetisch |
as_factor() |
forcats | Nach Auftreten im Vektor |
as_factor() ist oft praktischer, weil die Reihenfolge der Daten erhalten bleibt.
Levels explizit angeben
Mit dem levels-Argument können wir die Reihenfolge selbst bestimmen:
[1] mittel hoch niedrig hoch mittel
Levels: hoch mittel niedrig[1] mittel hoch niedrig hoch mittel
Levels: niedrig mittel hochforcats: Faktoren manipulieren
Das {forcats}-Paket (Teil des tidyverse) bietet praktische Funktionen zur Factor-Manipulation. Alle Funktionen beginnen mit fct_.
Reihenfolge ändern
fct_relevel() – Manuell umsortieren
[1] "blue" "blue-gray" "brown" "dark" "hazel" "unknown"
[7] "yellow" [1] "brown" "blue" "blue-gray" "dark" "hazel" "unknown"
[7] "yellow" [1] "brown" "blue" "hazel" "blue-gray" "dark" "unknown"
[7] "yellow" fct_reorder() – Nach anderer Variable sortieren
Besonders nützlich für Grafiken – sortiere Kategorien nach einem numerischen Wert:
# Durchschnittliche Größe pro Haarfarbe
hair_height <- humans %>%
filter(!is.na(hair_color), !is.na(height)) %>%
group_by(hair_color) %>%
summarise(mean_height = mean(height), n = n()) %>%
filter(n >= 2) # Nur Gruppen mit mindestens 2 Personen
# Ohne fct_reorder: alphabetisch
p1 <- hair_height %>%
ggplot(aes(x = hair_color, y = mean_height)) +
geom_col() +
labs(title = "Alphabetisch") +
theme_minimal() +
theme(axis.text.x = element_text(angle = 45, hjust = 1))
# Mit fct_reorder: nach Größe sortiert
p2 <- hair_height %>%
ggplot(aes(x = fct_reorder(hair_color, mean_height), y = mean_height)) +
geom_col() +
labs(title = "Nach Größe sortiert", x = "hair_color") +
theme_minimal() +
theme(axis.text.x = element_text(angle = 45, hjust = 1))
p1 + p2fct_infreq() – Nach Häufigkeit sortieren
[1] "brown" "blue" "hazel" "yellow" "blue-gray" "dark"
[7] "unknown" Die häufigsten Kategorien kommen zuerst – ideal für Balkendiagramme.
fct_rev() – Reihenfolge umkehren
Levels zusammenfassen
fct_lump_n() – Seltene in “Other” zusammenfassen
# Nur die 3 häufigsten behalten, Rest wird "Other"
humans %>%
mutate(eye_color = fct_lump_n(eye_color, n = 3)) %>%
tabyl(eye_color) eye_color n percent
blue 12 0.34285714
brown 16 0.45714286
hazel 2 0.05714286
yellow 2 0.05714286
Other 3 0.08571429# Mit deutschem Label
humans %>%
mutate(eye_color = fct_lump_n(eye_color, n = 3, other_level = "Sonstige")) %>%
tabyl(eye_color) eye_color n percent
blue 12 0.34285714
brown 16 0.45714286
hazel 2 0.05714286
yellow 2 0.05714286
Sonstige 3 0.08571429Verwandte Funktionen:
-
fct_lump_min()– Zusammenfassen wenn weniger als n Vorkommen -
fct_lump_prop()– Zusammenfassen wenn Anteil unter x%
fct_collapse() – Mehrere Levels zusammenfassen
Levels umbenennen
fct_recode()
humans %>%
mutate(
eye_color_de = fct_recode(
eye_color,
"Blau" = "blue",
"Braun" = "brown",
"Dunkel" = "dark",
"Haselnuss" = "hazel",
"Blau-Grau" = "blue-gray"
)
) %>%
tabyl(eye_color_de) eye_color_de n percent
Blau 12 0.34285714
Blau-Grau 1 0.02857143
Braun 16 0.45714286
Dunkel 1 0.02857143
Haselnuss 2 0.05714286
unknown 1 0.02857143
yellow 2 0.05714286Arbeite mit dem humans-Datensatz:
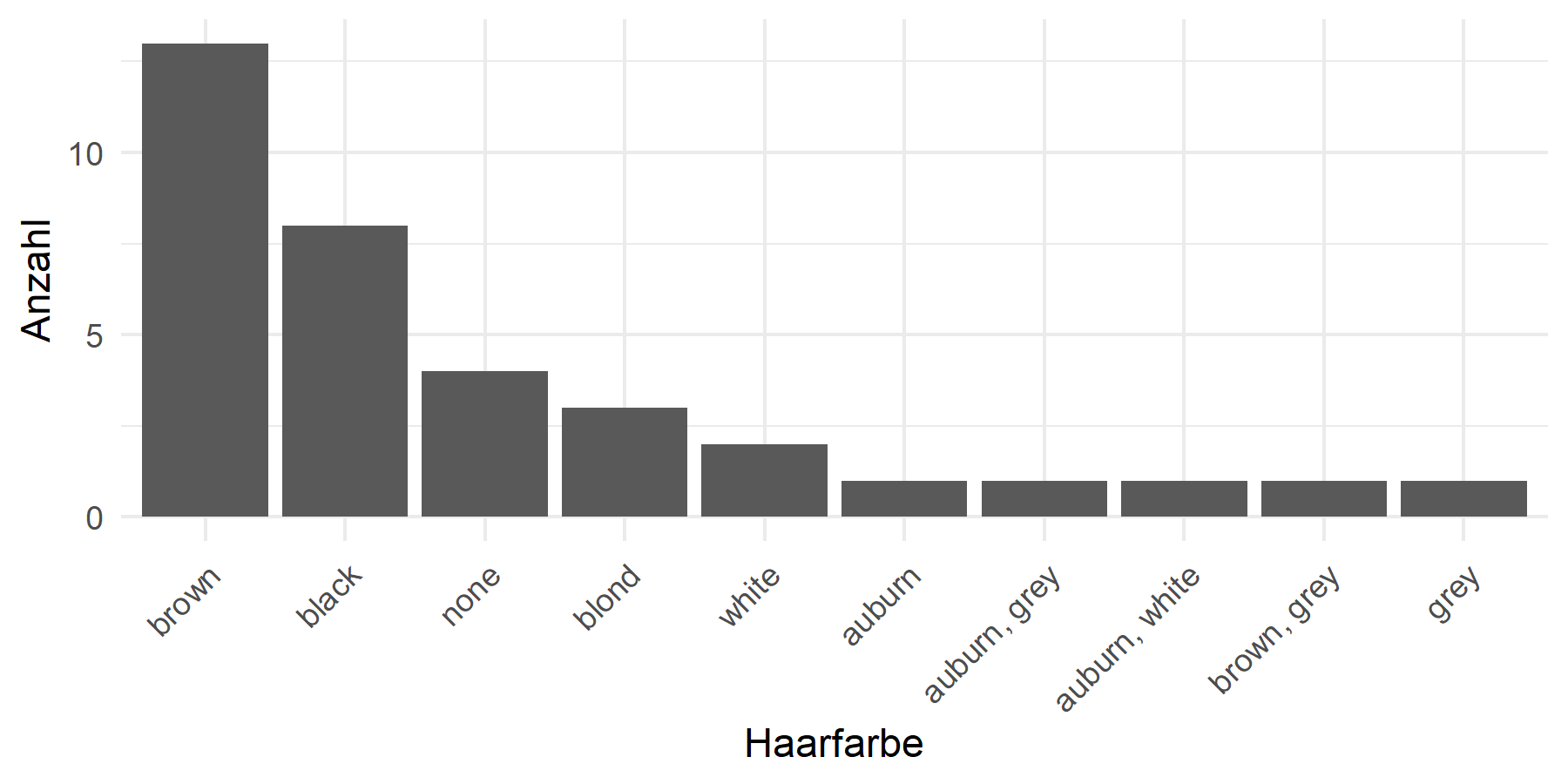
a) Erstelle ein Balkendiagramm von hair_color, bei dem die Balken nach Häufigkeit sortiert sind (häufigste links).
b) Fasse alle Haarfarben außer den 3 häufigsten unter “Andere” zusammen und erstelle eine Häufigkeitstabelle mit tabyl().
c) Benenne die Levels von gender um: “feminine” → “weiblich”, “masculine” → “männlich”.
# a) Balkendiagramm nach Häufigkeit
humans %>%
filter(!is.na(hair_color)) %>%
ggplot(aes(x = fct_infreq(hair_color))) +
geom_bar() +
labs(x = "Haarfarbe", y = "Anzahl") +
theme_minimal() +
theme(axis.text.x = element_text(angle = 45, hjust = 1))# b) Lumpen und tabyl
humans %>%
mutate(hair_color = fct_lump_n(hair_color, n = 3, other_level = "Andere")) %>%
tabyl(hair_color, show_na = FALSE) hair_color n percent
black 8 0.2285714
brown 13 0.3714286
none 4 0.1142857
Andere 10 0.2857143# c) Gender umbenennen
humans %>%
mutate(
gender_de = fct_recode(
gender,
"weiblich" = "feminine",
"männlich" = "masculine"
)
) %>%
tabyl(gender_de) gender_de n percent
weiblich 9 0.2571429
männlich 26 0.7428571Praktische Anwendungen
Faktoren in ggplot2
Die Level-Reihenfolge bestimmt die Anordnung in Grafiken:
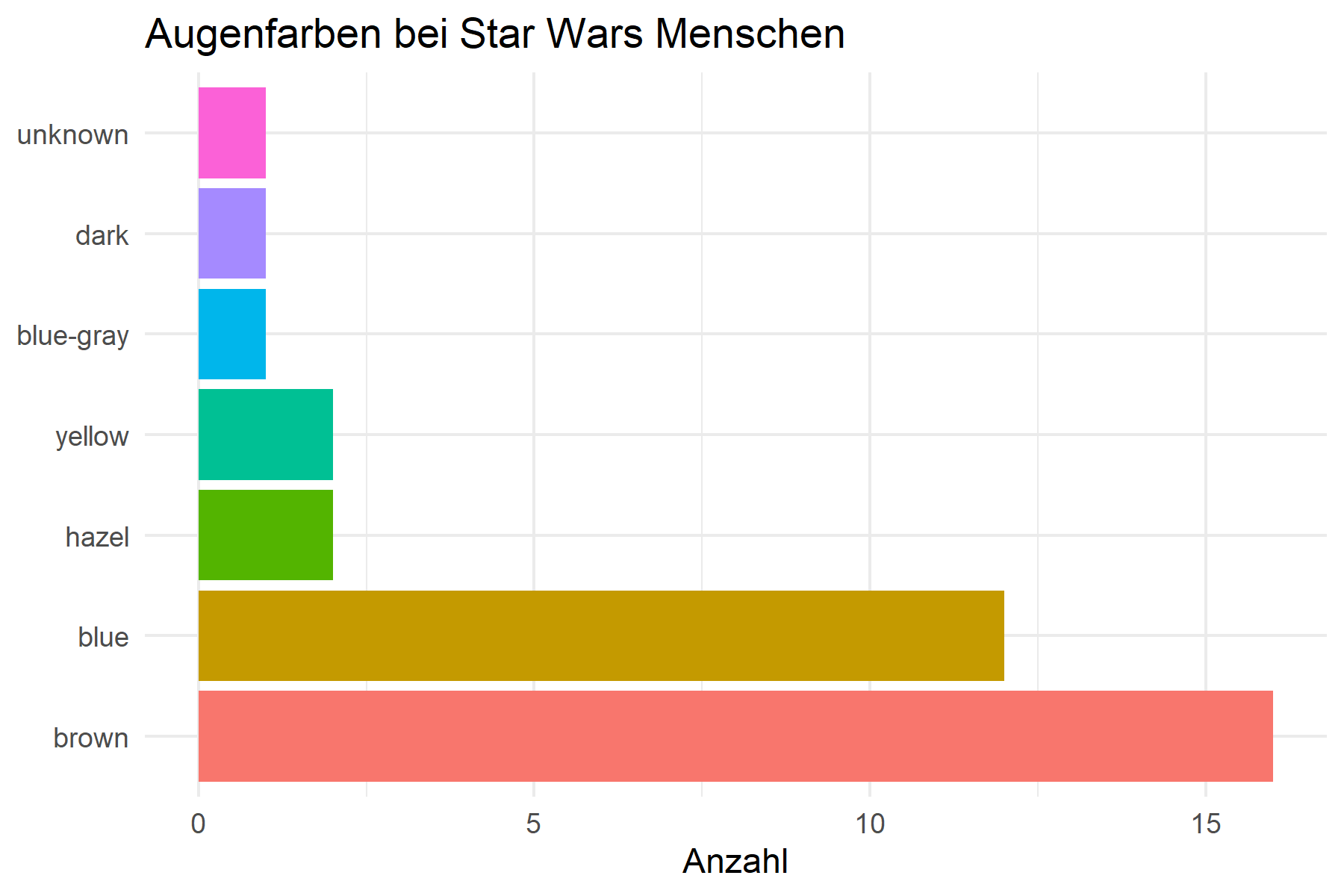
# Sinnvolle Reihenfolge für Balkendiagramm
humans %>%
filter(!is.na(eye_color)) %>%
mutate(eye_color = fct_infreq(eye_color)) %>% # Nach Häufigkeit
ggplot(aes(x = eye_color, fill = eye_color)) +
geom_bar() +
coord_flip() + # Horizontale Balken
labs(
title = "Augenfarben bei Star Wars Menschen",
x = NULL,
y = "Anzahl"
) +
theme_minimal() +
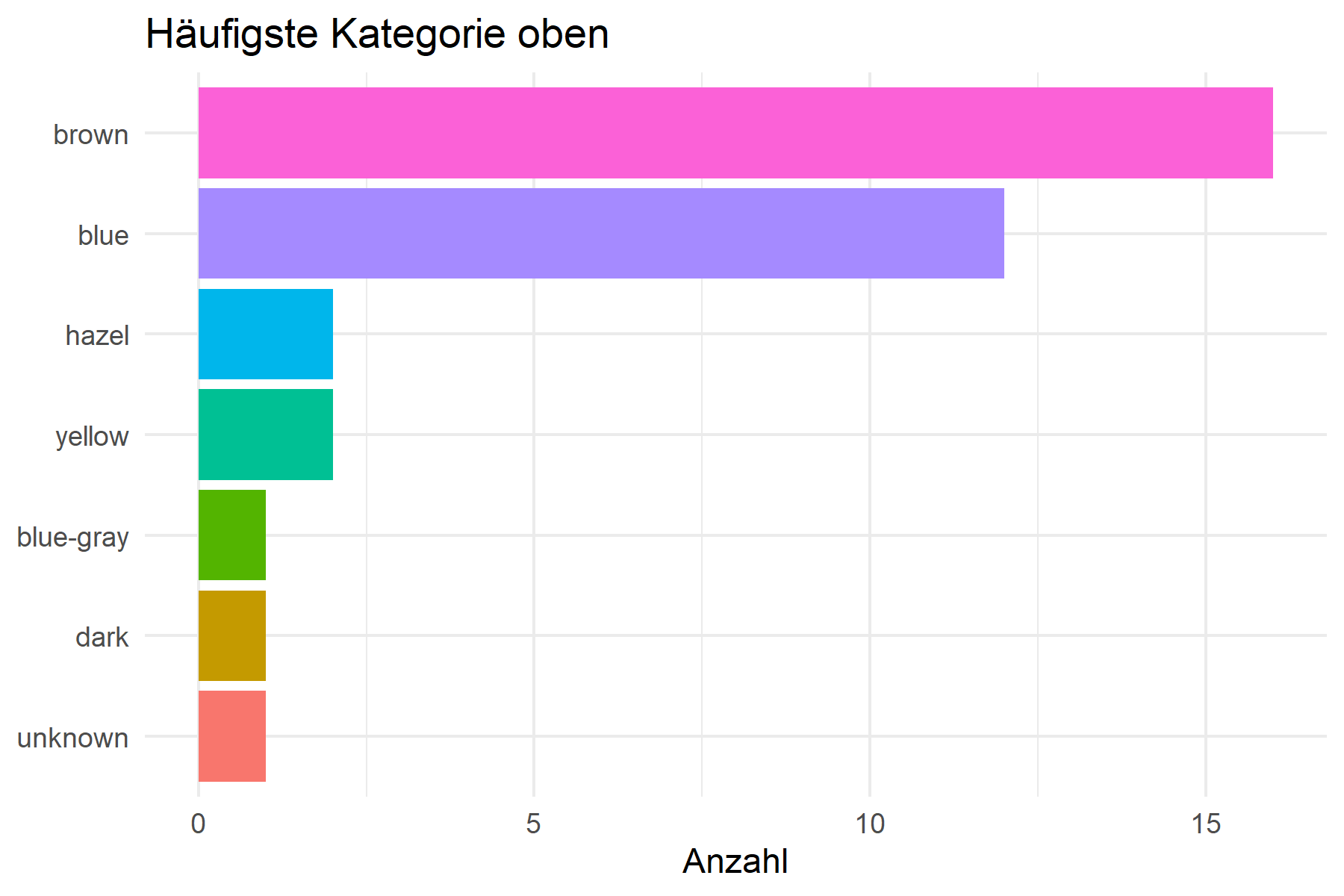
theme(legend.position = "none")Bei horizontalen Balkendiagrammen will man oft die häufigste Kategorie oben haben. Dafür kombiniert man fct_infreq() mit fct_rev():
humans %>%
filter(!is.na(eye_color)) %>%
mutate(eye_color = fct_rev(fct_infreq(eye_color))) %>% # Umgekehrt
ggplot(aes(x = eye_color, fill = eye_color)) +
geom_bar() +
coord_flip() +
labs(
title = "Häufigste Kategorie oben",
x = NULL,
y = "Anzahl"
) +
theme_minimal() +
theme(legend.position = "none")Faktoren in tabyl()
Auch tabyl() respektiert die Level-Reihenfolge:
eye_color n percent
blue 12 0.34285714
blue-gray 1 0.02857143
brown 16 0.45714286
dark 1 0.02857143
hazel 2 0.05714286
unknown 1 0.02857143
yellow 2 0.05714286# Mit Factor: nach unserer Reihenfolge
humans %>%
mutate(eye_color = fct_relevel(eye_color, "brown", "blue")) %>%
tabyl(eye_color) eye_color n percent
brown 16 0.45714286
blue 12 0.34285714
blue-gray 1 0.02857143
dark 1 0.02857143
hazel 2 0.05714286
unknown 1 0.02857143
yellow 2 0.05714286# Nach Häufigkeit
humans %>%
mutate(eye_color = fct_infreq(eye_color)) %>%
tabyl(eye_color) eye_color n percent
brown 16 0.45714286
blue 12 0.34285714
hazel 2 0.05714286
yellow 2 0.05714286
blue-gray 1 0.02857143
dark 1 0.02857143
unknown 1 0.02857143Dies ist besonders nützlich für Berichte, wo die Reihenfolge der Kategorien eine inhaltliche Bedeutung haben soll (z.B. “sehr gut”, “gut”, “befriedigend”, “schlecht”).
Zusammenfassung
Faktoren sind mächtiger als einfache Character-Variablen, weil sie eine definierte Menge an Kategorien mit einer bestimmten Reihenfolge speichern.
Character vs. Factor:
| Aspekt | Character | Factor |
|---|---|---|
| Speichert | Nur Text | Text + Levels + Reihenfolge |
| Sortierung | Alphabetisch | Nach Level-Reihenfolge |
| Unbekannte Werte | Erlaubt | Werden NA (wenn nicht in Levels) |
| Anwendung | Freitext | Kategorien mit fester Ausprägung |
Wann Character, wann Factor?
- Character: Freitext, Namen, IDs, Kommentare
- Factor: Kategorien mit definierter Ausprägung (Geschlecht, Likert-Skalen, Regionen)
Die wichtigsten forcats-Funktionen:
| Funktion | Zweck |
|---|---|
fct_relevel() |
Levels manuell umsortieren |
fct_reorder() |
Nach numerischer Variable sortieren |
fct_infreq() |
Nach Häufigkeit sortieren |
fct_rev() |
Reihenfolge umkehren |
fct_lump_n() |
Seltene zu “Other” zusammenfassen |
fct_collapse() |
Mehrere Levels zusammenfassen |
fct_recode() |
Levels umbenennen |
Typischer Workflow für Grafiken:
Weiterführende Ressourcen:
Zitat
@online{schmidt2026,
author = {{Dr. Paul Schmidt}},
publisher = {BioMath GmbH},
title = {5. Faktoren},
date = {2026-02-07},
url = {https://biomathcontent.netlify.app/de/content/r_more/05_factors.html},
langid = {de}
}